大数据知识图谱——基于知识图谱+深度学习的大数据NLP医疗知识问答可视化系统(全网最详细讲解及源码/建议收藏)
一、项目概述
知识图谱是将知识连接起来形成的一个网络。由节点和边组成,节点是实体,边是两个实体的关系,节点和边都可以有属性。知识图谱除了可以查询实体的属性外,还可以很方便的从一个实体通过遍历关系的方式找到相关的实体及属性信息。
基于知识图谱+flask的KBQA医疗问答系统基于医疗方面知识的问答,通过搭建一个医疗领域知识图谱,并以该知识图谱完成自动问答与分析服务。 基于知识图谱+flask的KBQA医疗问答系统以neo4j作为存储,本系统知识图谱建模使用的最大向前匹配是一种贪心算法,从句首开始匹配,每次选择最长的词语。由于只需一次遍历,因此在速度上相对较快。 算法相对简单,容易实现和理解,不需要复杂的数据结构。 对于中文文本中大部分是左向的情况,最大向前匹配通常能够较好地切分。与最大向前匹配相反,最大向后匹配从句尾开始匹配,每次选择最长的词语。适用于大部分右向的中文文本。双向最大匹配结合了最大向前匹配和最大向后匹配的优势,从两个方向分别匹配,然后选择分词数量较少的一种结果。这种方法综合考虑了左向和右向的特点,提高了切分的准确性,以关键词执行cypher查询,并返回相应结果查询语句作为问答。后面我又设计了一个简单的基于 Flask 的聊天机器人应用,利用nlp自然语言处理,通过医疗AI助手根据用户的问题返回结果,用户输入和系统返回的输出结果都会一起自动存储到sql数据库。后面又封装了深度学习模型完成一个完整知识图谱问答可视化系统。
具体详情见另一篇文章:大数据知识图谱之深度学习——基于BERT+LSTM+CRF深度学习识别模型医疗知识图谱问答可视化系统
在多模式匹配方面, Aho-Corasick算法专门用于在一个文本中同时搜索多个模式(关键词)。相比于暴力搜索算法,Aho-Corasick算法的时间复杂度较低,在本知识图谱建模问答系统中,性能更为显著。在线性时间复杂度方面,进行预处理的阶段,Aho-Corasick算法构建了一个确定性有限自动机(DFA),使得在搜索阶段的时间复杂度为O(n),其中n是待搜索文本的长度。这种线性时间复杂度使得算法在本应用中非常高效。在灵活性方面, Aho-Corasick算法在构建有限自动机的过程中,可以方便地添加、删除模式串,而不需要重新构建整个数据结构,提高了算法的灵活性和可维护性。在医疗领域中,针对与医疗相关的疾病、症状、药物、问诊等内容进行系统性的内容定义,也能够通过命名实体识别来对药剂、价格、收款等内容进行定义,这种定义的方式可以通过精准匹配的方式来进行实体边界的识别,并且可以实现对边界的正确标记。命名实体识别的方法主要有三种方式,第一种是通过利用规则法来进行规则的人工编写;第二种是通过HMM、CRF等模型来进行机器学习模板订制;第三种是通过神经网络的方式以LSTM、RNN等算法来进行特征的提取。
二、实现知识图谱的医疗知识问答系统基本流程
-
配置好所需要的环境(jdk,neo4j,pycharm,python等)
-
爬取所需要的医学数据,获取所需基本的医疗数据。
-
对医疗数据进行数据清洗处理。
-
基于贪心算法进行分词策略。
-
关系抽取定义与实体识别。
-
知识图谱建模。
-
基于
Aho-Corasick算法进行多模式匹配。 -
设计一个基于 Flask 的聊天机器人AI助手。
-
设计用户输入和系统输出记录数据自动存储到sql数据库。
-
封装深度学习模型完整系统——基于BERT+LSTM+CRF深度学习识别模型医疗知识图谱问答可视化系统
三、项目工具所用的版本号
Neo4j版本:Neo4j Desktop1.4.15;
neo4j里面医疗系统数据库版本:4.4.5;
Pycharm版本:2021;
JDK版本:jdk1.8.0_211;
MongoDB版本:MongoDB-windows-x86_64-5.0.14;
flask版本:3.0.0
Django版本:3.2.3
四、所需要软件的安装和使用
(一)安装JAVA
1.下载java安装包:
官网下载链接:https://www.oracle.com/java/technologies/javase-downloads.html
本人下载的版本为JDK-1.8,JDK版本的选择一定要恰当,版本太高或者太低都可能导致后续的neo4j无法使用。
安装好JDK之后就要开始配置环境变量了。 配置环境变量的步骤如下:
右键单击此电脑—点击属性—点击高级系统设置—点击环境变量
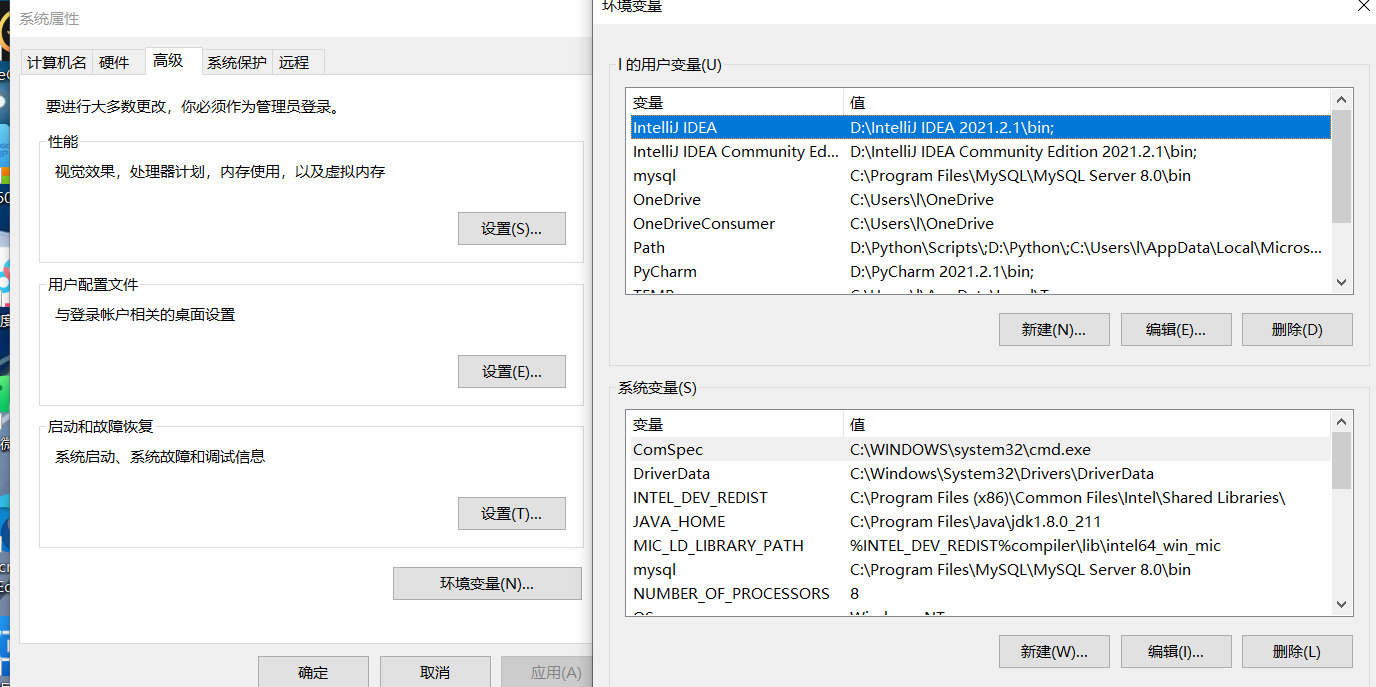
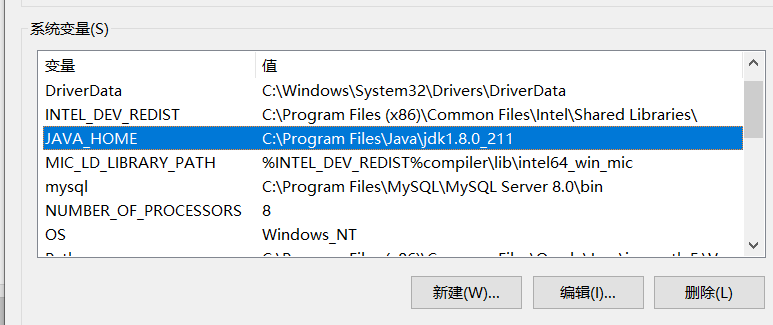
在下方的系统变量区域,新建环境变量,命名为JAVA_HOME,变量值设置为刚才JAVA的安装路径,我这里是C:\Program Files\Java\jdk1.8.0_211
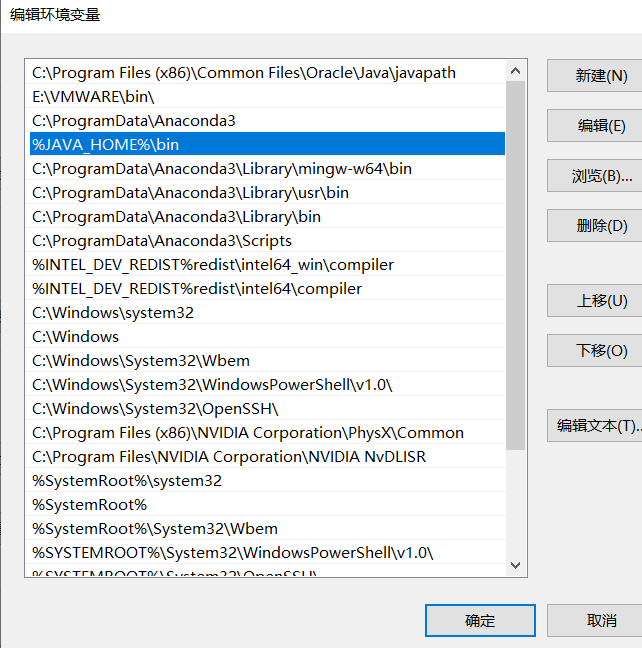
编辑系统变量区的Path,点击新建,然后输入 %JAVA_HOME%\bin
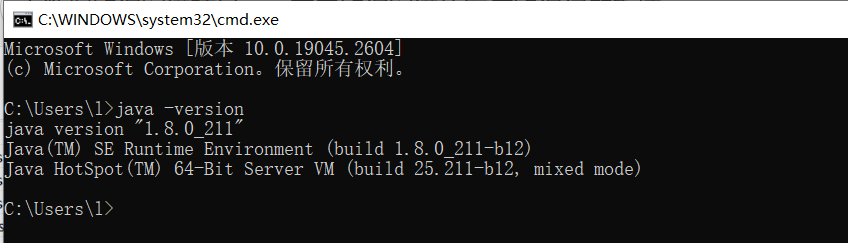
打开命令提示符CMD(WIN+R,输入cmd),输入 java -version,若提示Java的版本信息,则证明环境变量配置成功。
2.安装好JDK之后,就可以安装neo4j了
2.1 下载neo4j
官方下载链接:https://neo4j.com/download-center/
也可以直接下载我上传到云盘链接:
Neo4j Desktop Setup 1.4.15.exe
链接:https://pan.baidu.com/s/1eXw0QjqQ9nfpXwR09zLQHA?pwd=2023
提取码:2023
打开之后会有一个自己设置默认路径,可以根据自己电脑情况自行设置,然后等待启动就行了
打开之后我们新建一个数据库,名字叫做:“基于医疗领域的问答系统”
详细信息看下图:
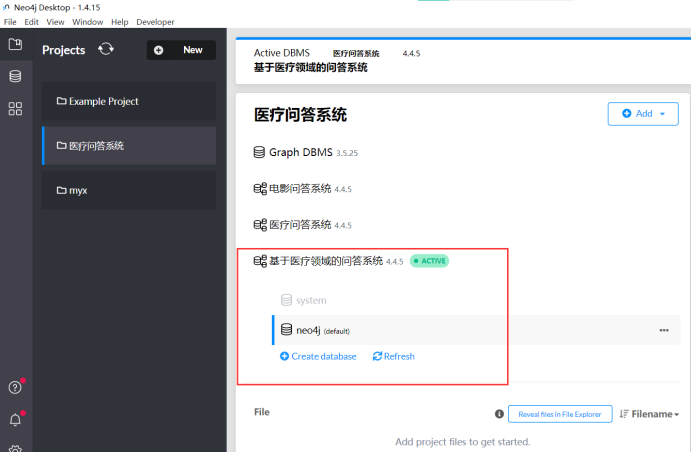
数据库所用的是4.4.5版本,其他数据库参数信息如下:

五、项目结构整体目录
├── pycache \\编译结果的保存目录
│ │ ├── answer_search.cpython-39.pyc
│ │ ├── question_classifier.cpython-39.pyc
│ │ │── 基于问句解析.cpython-39.pyc
│ │ │── 基于问句解析结果查询.cpython-39.pyc
│ │ │── 基于问题分类.cpython-39.pyc
├── data\本项目的数据
│ └── medical.json \本项目的数据,通过build_medicalgraph.py导neo4j
├── dict
│ ├── check.txt \诊断检查项目实体库
│ ├── deny.txt \否定词词库
│ ├── department.txt \医疗科目实体库
│ ├── disease.txt \疾病实体库
│ ├── drug.txt \药品实体库
│ ├── food.txt \食物实体库
│ ├── producer.txt \在售药品库
│ └── symptom.txt \疾病症状实体库
├── prepare_data \爬虫及数据处理
│ ├──__pycache__
│ ├── build_data.py \数据库操作脚本
│ ├── data_spider.py \网络资讯采集脚本
│ └── max_cut.py \基于词典的最大向前/向后脚本
│ ├── 处理后的medical数据.xlsx
│ ├── MongoDB数据转为json格式数据文件.py
│ ├──从MongoDB导出的medical.csv
│ ├──data.json #从MongoDB导出的json格式数据
├── static \静态资源文件
├── templates
│ ├──index.html \问答系统前端UI页面
├── app.py \启动flask问答AI脚本主程序
├── question_classifier.py \问句类型分类脚本
├── answer_search.py \基于问题答复脚本
├── build_medicalgraph.py \构建医疗图谱脚本
├── chatbot_graph.py \医疗AI助手问答系统机器人脚本
├── question_parser.py [\基于问句解析脚本](file://基于问句解析脚本)
├── 删除所有关系.py \可有可无的脚本文件
├── 删除关系链.py \可有可无的脚本文件
├── 两节点新加关系.py \可有可无的脚本文件
├── 交互 匹配所有节点.py \可有可无的脚本文件
这里chatbot_graph.py脚本首先从需要运行的chatbot_graph.py文件开始分析。
该脚本构造了一个问答类ChatBotGraph,定义了QuestionClassifier类型的成员变量classifier、QuestionPase类型的成员变量parser和AnswerSearcher类型的成员变量searcher。
question_classifier.py脚本构造了一个问题分类的类QuestionClassifier,定义了特征词路径、特征词、领域actree、词典、问句疑问词等成员变量。question_parser.py问句分类后需要对问句进行解析。该脚本创建一个QuestionPaser类,该类包含三个成员函数。
answer_search.py问句解析之后需要对解析后的结果进行查询。该脚本创建了一个AnswerSearcher类。与build_medicalgraph.py类似,该类定义了Graph类的成员变量g和返回答案列举的最大个数num_list。该类的成员函数有两个,一个查询主函数一个回复模块。
问答系统框架的构建是通过chatbot_graph.py、answer_search.py、question_classifier.py、question_parser.py等脚本实现。
五、系统实现
数据的抓取与存储
安装MongoDB数据库:
MongoDB官方下载地址:
https://www.mongodb.com/try

安装完成:
然后我们进行MongoDB的环境配置:
在变量Path里加入E:\MongoDB\bin
打开终端(cmd)输入mongod --dbpath E:\MongoDB\data\db
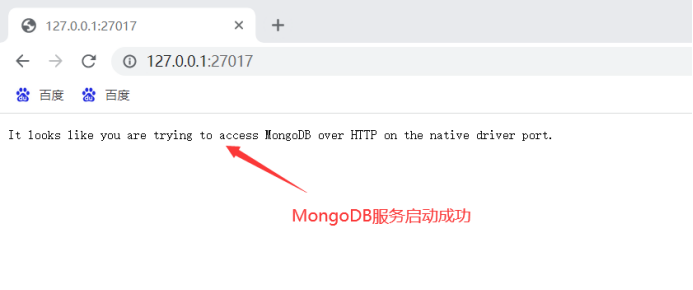
在浏览器输入
127.0.0.1:27017 查看MongoDB服务是否启动成功:
安装好MongoDB之后开始爬取数据:
数据来源于寻医问药网:http://jib.xywy.com/
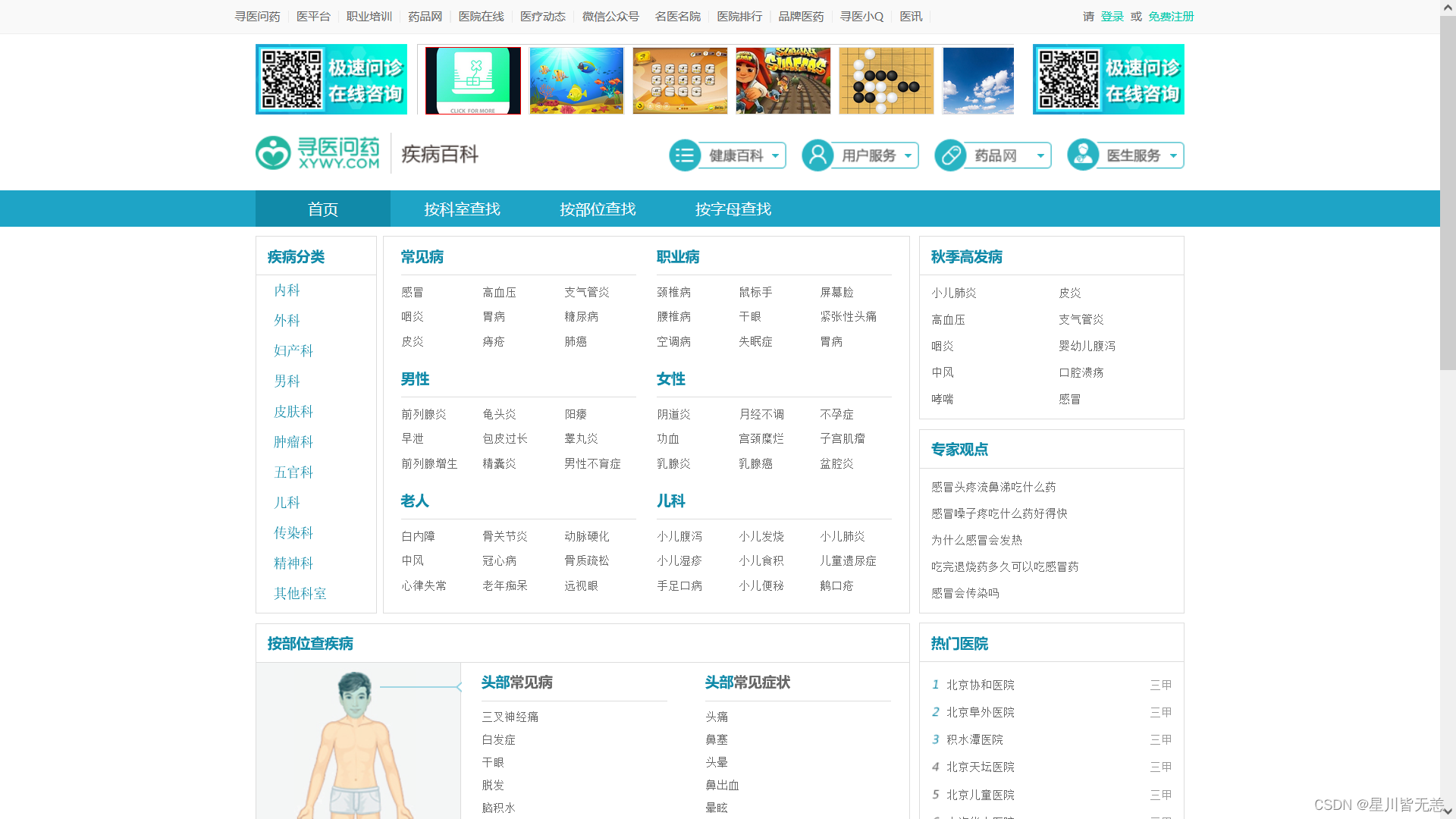
具体的疾病详情页面如下:
首先对网址上的疾病链接进行分析,以感冒为例:
感冒的链接:http://jib.xywy.com/il_sii_38.htm
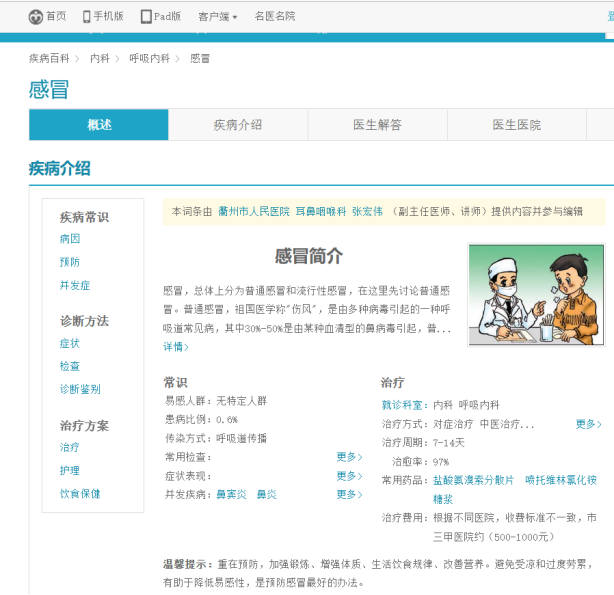
可以看到,上面包含了疾病的简介、病因、预防、症状、检查、治疗、并发症、饮食保健等详情页的内容。下面我们要使用爬虫把信息收集起来。要收集 url 下面对应的数据,具体爬虫代码如下:
之前老版本的insert方法被弃用,再用会出现警告。insert 替换为了 insert_one,这样就不会再收到关于 insert 方法被弃用的警告了。
'''基于寻医问药的医疗数据采集''' # 使用 insert_one 或 insert_many 方法。提供了更多的灵活性,并且支持更多的功能,比如插入后返回的文档的 _id 值。 class MedicalSpider: def __init__(self): self.conn = pymongo.MongoClient() self.db = self.conn['medical2'] self.col = self.db['data'] def insert_data(self, data): # 使用 insert_one 方法插入单个文档 self.col.insert_one(data) 根据url,请求html
用于获取指定 URL 的 HTML 内容的函数,使用了 Python 的 requests 库。在这个代码中,设置请求的头部信息(User-Agent),代理信息(proxies),然后使用 requests.get 方法获取页面内容,并指定了编码为 ‘gbk’。
'''根据url,请求html''' def get_html(self,url): headers = { 'user-agent': 'Mozilla/5.0 (Windows NT 10.0; WOW64) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/69.0.3497.100 Safari/537.36' } proxies = { 'http': None, 'https': None } html = requests.get(url=url, headers=headers, proxies=proxies) html.encoding = 'gbk' return html.text 爬取医疗相关的信息,包括疾病描述、疾病预防、疾病症状、治疗方法等。
url_parser 函数
def url_parser(self, content): selector = etree.HTML(content) urls = ['http://www.anliguan.com' + i for i in selector.xpath('//h2[@class="item-title"]/a/@href')] return urls url_parser 函数函数的作用是解析传入的 HTML 内容,提取出页面中疾病相关的链接。具体来说:
etree.HTML(content): 使用 lxml 库中的 etree 模块将 HTML 内容转换成可被 XPath 解析的对象。selector.xpath('//h2[@class="item-title"]/a/@href'): 使用 XPath 选择器提取所有<h2>标签中class属性为"item-title"的子节点<a>的href属性,得到的是一组相对链接。['http://www.anliguan.com' + i for i in ...]: 将相对链接转换成完整的链接,拼接在'http://www.anliguan.com'前面。
这个函数的作用是解析传入的 HTML 内容,提取出页面中疾病相关的链接。具体来说:
spider_main函数是主要的爬虫逻辑,循环遍历页面,爬取不同类型的医疗信息,并将结果存储到数据库中
'''url解析''' def url_parser(self,content): selector=etree.HTML(content) urls=['http://www.anliguan.com'+i for i in selector.xpath('//h2[@class="item-title"]/a/@href')] return urls '''主要的爬取的链接''' def spider_main(self): # 收集页面 for page in range(1,11000): try: basic_url='http://jib.xywy.com/il_sii/gaishu/%s.htm'%page # 疾病描述 cause_url='http://jib.xywy.com/il_sii/cause/%s.htm'%page # 疾病起因 prevent_url='http://jib.xywy.com/il_sii/prevent/%s.htm'%page # 疾病预防 symptom_url='http://jib.xywy.com/il_sii/symptom/%s.htm'%page #疾病症状 inspect_url='http://jib.xywy.com/il_sii/inspect/%s.htm'%page # 疾病检查 treat_url='http://jib.xywy.com/il_sii/treat/%s.htm'%page # 疾病治疗 food_url = 'http://jib.xywy.com/il_sii/food/%s.htm' % page # 饮食治疗 drug_url = 'http://jib.xywy.com/il_sii/drug/%s.htm' % page #药物 basic_url、symptom_url、food_url、drug_url和cause_url是根据page变量构建的不同 URL 地址,用于访问和爬取不同类型信息的网页。
-data是一个字典,用于存储从不同 URL 中获取的数据。
2. 对不同 URL 进行爬取和信息提取:
-data['basic_info'] = self.basicinfo_spider(basic_url):调用basicinfo_spider方法,从basic_url获取基础信息,并将其存储在data字典的'basic_info'键下。
-data['symptom_info'] = self.symptom_spider(symptom_url):类似地,获取症状信息并存储在'symptom_info'键下。
-data['food_info'] = self.food_spider(food_url):获取饮食信息并存储在'food_info'键下。
-data['drug_info'] = self.drug_spider(drug_url):获取药物信息并存储在'drug_info'键下。
-data['cause_info'] = self.cause_spider(cause_url):获取病因信息并存储在'cause_info'键下。
3. 打印和数据库存储:
-print(page, basic_url):打印当前页面和基础 URL,用于调试和跟踪程序执行过程。
-print(data):打印从不同 URL 获取的数据,同样用于调试和查看程序执行结果。
-self.col.insert_one(data):将获取的数据存入 MongoDB 数据库中。
4. 异常处理:
-except Exception as e::捕获所有类型的异常,并将异常对象赋值给变量e。
-print(e, page):在捕获到异常时打印异常信息和页面信息,以便于调试和追踪出错的原因。
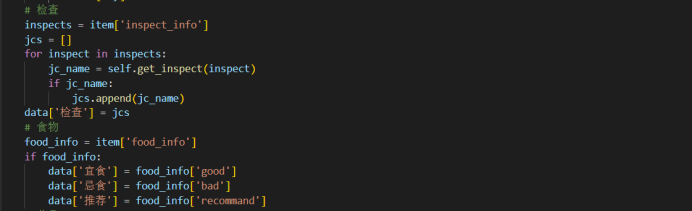
data={} data['url'] = basic_url data['basic_info'] = self.basicinfo_spider(basic_url) data['cause_info'] = self.common_spider(cause_url) data['prevent_info'] = self.common_spider(prevent_url) data['symptom_info'] = self.symptom_spider(symptom_url) data['inspect_info'] = self.inspect_spider(inspect_url) data['treat_info'] = self.treat_spider(treat_url) data['food_info'] = self.food_spider(food_url) data['drug_info'] = self.drug_spider(drug_url) print(page, basic_url) self.col.insert_one(data) except Exception as e: print(e,page) 需要爬取的信息包括疾病名、所属目录、症状、治疗方案等等,都可以从页面上获取。
MongoDB里面的数据也是刷新显示最新数据记录
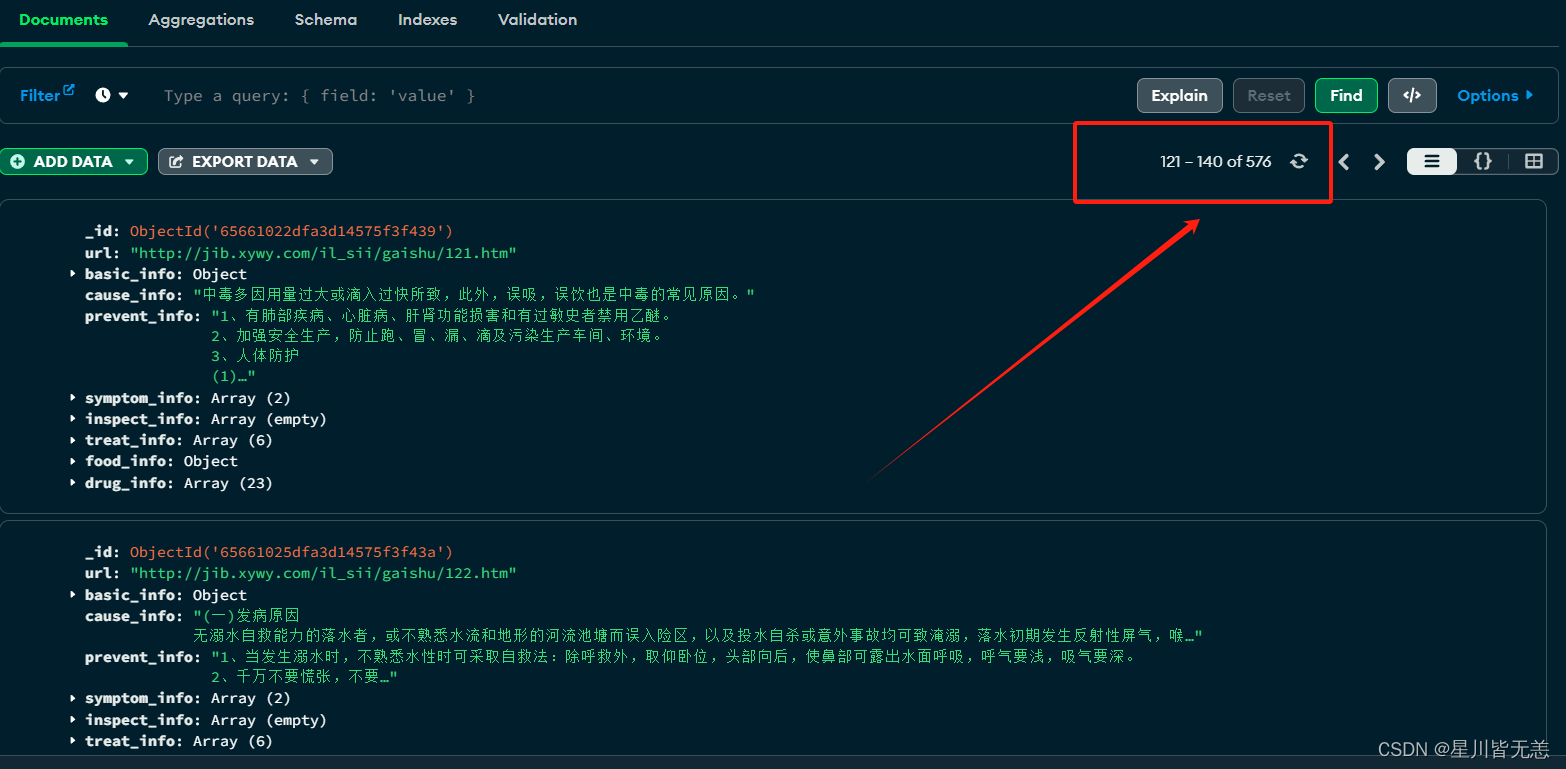
我们随便点一个我们爬取的网页链接,点击查看网页详情:
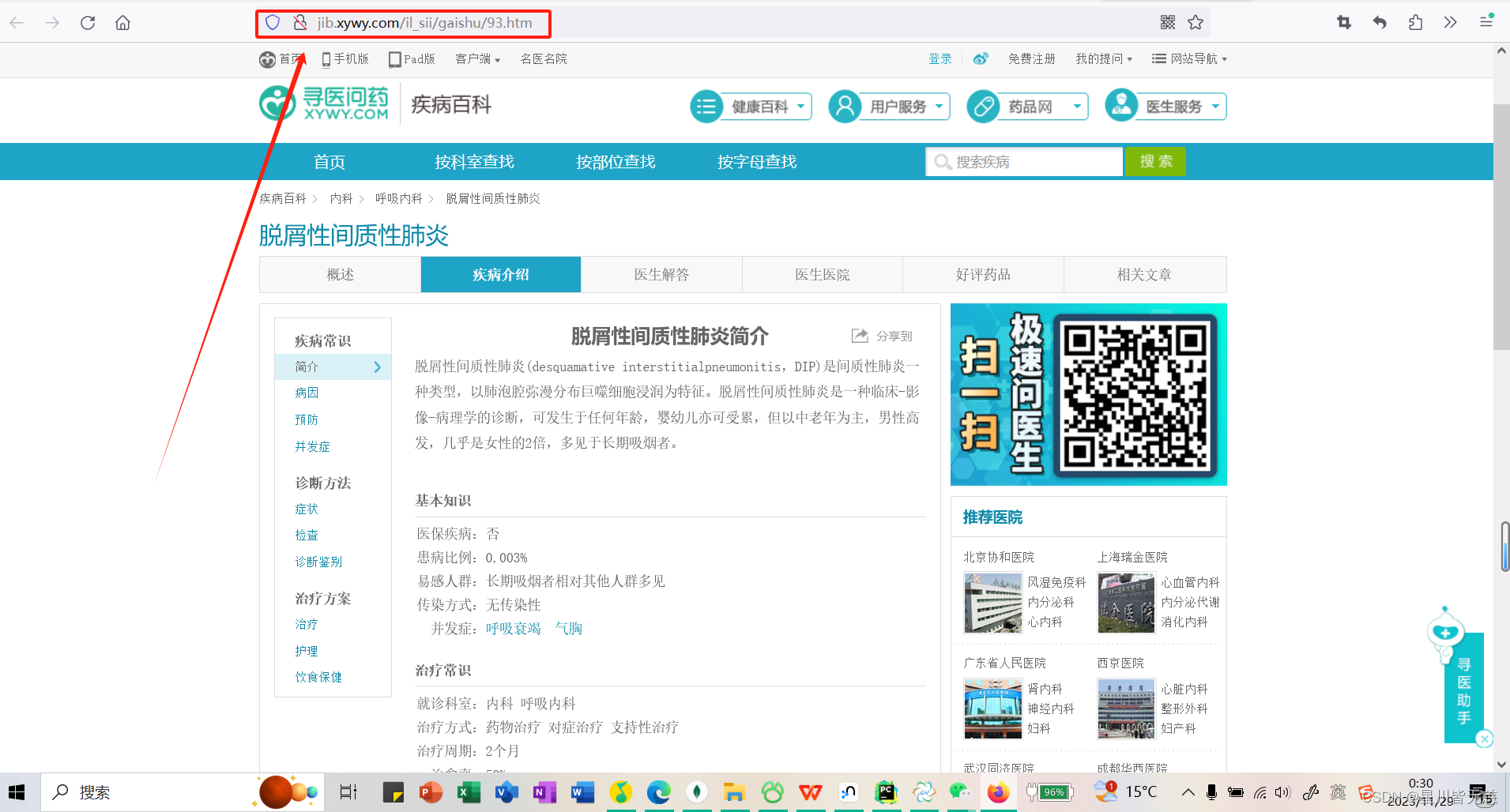
综上内容所述,这里做一个总结
这里代码爬虫的主要功能是爬取疾病相关的信息,并将数据存储到MongoDB数据库中。代码的主要结构是一个名为MedicalSpider的类,它包含了各种方法来处理不同类型的数据采集任务。在代码的开头,导入了一些必要的库,如requests、urllib、lxml和pymongo。然后定义了一个MedicalSpider类,该类的构造函数初始化了MongoDB的连接,并指定了要使用的数据库和集合。
接下来是一系列方法,用于实现不同类型的数据采集。其中,get_html方法用于发送HTTP请求并获取网页的HTML内容。url_parser方法用于解析HTML内容,提取出需要的URL。basicinfo_spider方法用于解析疾病的基本信息,如名称、描述和所属目录。treat_spider、drug_spider和food_spider方法分别用于解析治疗信息、药物信息和食物信息。symptom_spider方法用于解析疾病的症状信息。inspect_spider方法用于解析疾病的检查信息。common_spider方法用于解析通用模块下的内容,如疾病预防和疾病起因。
在spider_main方法中,通过循环遍历页面,构造不同类型的URL,并调用相应的方法进行数据采集。采集到的数据以字典的形式存储,并插入到MongoDB数据库中。
最后,代码调用了MedicalSpider类的实例,并依次调用了inspect_crawl和spider_main方法,完成了数据的采集和存储。
通过爬取寻医问药网站的相关页面,获取疾病的基本信息、治疗信息、药物信息、食物信息、症状信息和检查信息,并将数据存储到MongoDB数据库中。
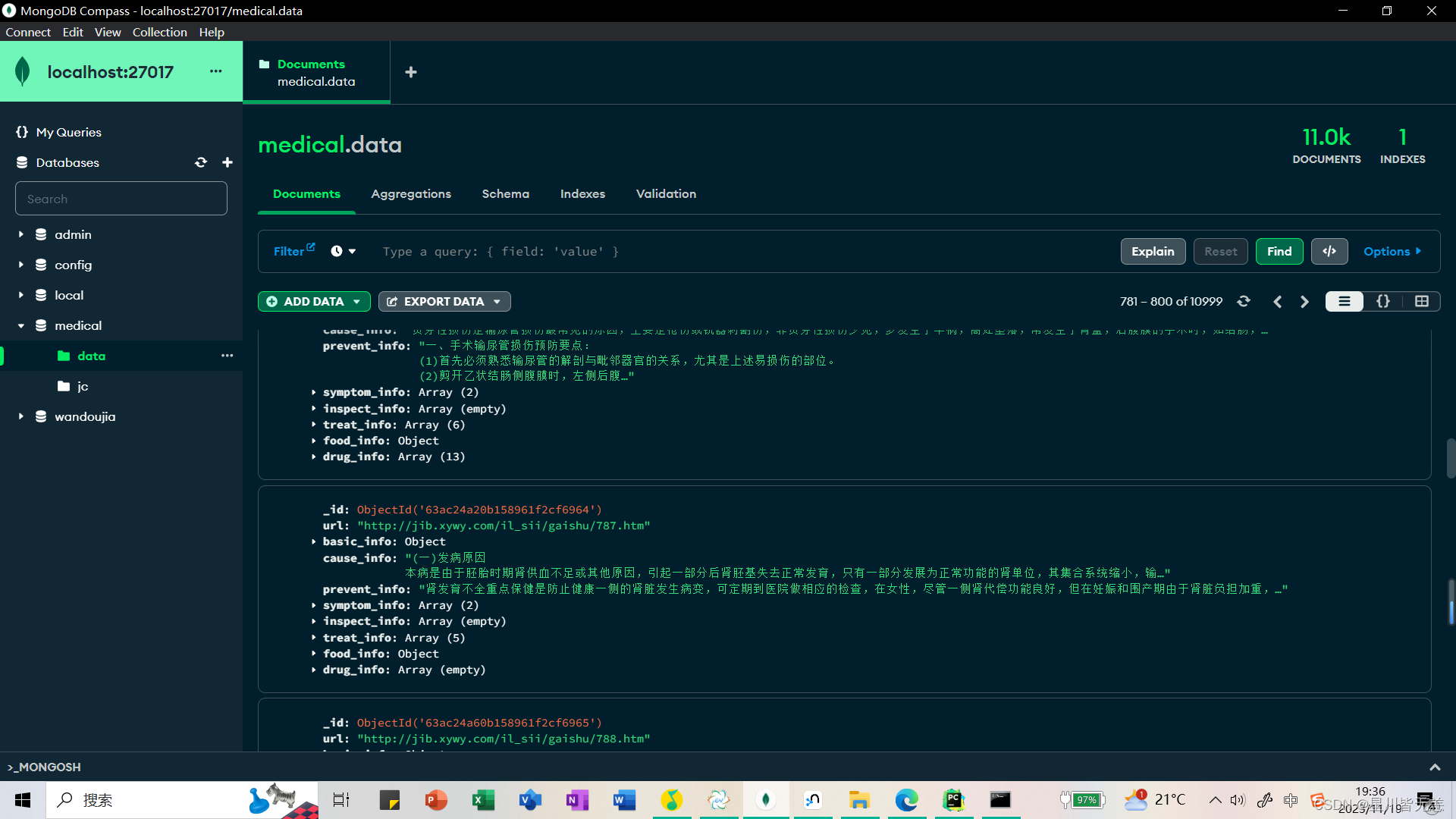
结束之后我们可以在MongoDB数据库中查看我们爬取到的疾病链接和解析出的网页内容:
贪心算法策略
贪心算法(又称贪婪算法)是指,在对问题求解时,总是做出在当前看来是最好的选择。也就是说,不从整体最优上加以考虑,他所做出的仅是在某种意义上的局部最优解。贪心算法不是对所有问题都能得到整体最优解,但对范围相当广泛的许多问题他能产生整体最优解或者是整体最优解的近似解。
贪婪算法(Greedy algorithm)是一种对某些求最优解问题的更简单、更迅速的设计技术。用贪婪法设计算法的特点是一步一步地进行,常以当前情况为基础根据某个优化测度作最优选择,而不考虑各种可能的整体情况,它省去了为找最优解要穷尽所有可能而必须耗费的大量时间,它采用自顶向下,以迭代的方法做出相继的贪心选择,每做一次贪心选择就将所求问题简化为一个规模更小的子问题,通过每一步贪心选择,可得到问题的一个最优解,虽然每一步上都要保证能获得局部最优解,但由此产生的全局解有时不一定是最优的,所以贪婪法不要回溯。
贪婪算法是一种改进了的分级处理方法。其核心是根据题意选取一种量度标准。然后将这多个输入排成这种量度标准所要求的顺序,按这种顺序一次输入一个量。如果这个输入和当前已构成在这种量度意义下的部分最佳解加在一起不能产生一个可行解,则不把此输入加到这部分解中。这种能够得到某种量度意义下最优解的分级处理方法称为贪婪算法。
对于一个给定的问题,往往可能有好几种量度标准。初看起来,这些量度标准似乎都是可取的,但实际上,用其中的大多数量度标准作贪婪处理所得到该量度意义下的最优解并不是问题的最优解,而是次优解。因此,选择能产生问题最优解的最优量度标准是使用贪婪算法的核心。
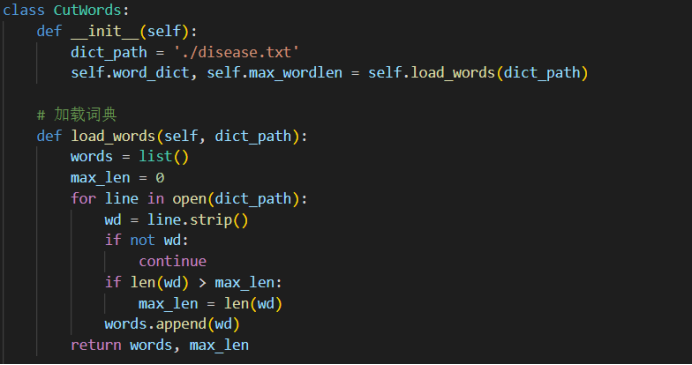
这里是用于中文分词的工具类,名为CutWords。它实现了最大向前匹配、最大向后匹配和双向最大匹配三种分词算法。在类的构造函数中,它首先初始化了一个词典路径,并调用load_words方法加载词典。load_words方法读取词典文件,将词语存储到一个列表中,并记录词语的最大长度。
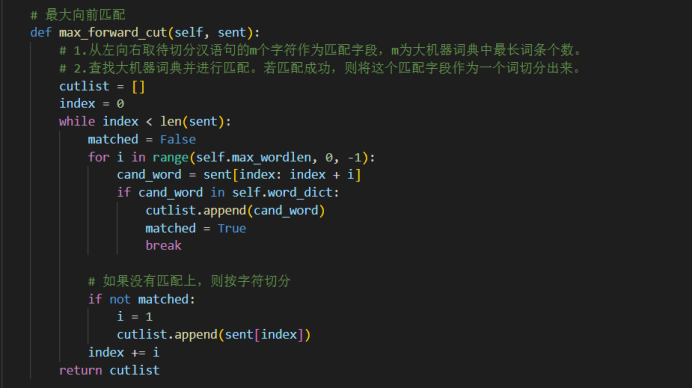
max_forward_cut方法实现了最大向前匹配算法。它从左向右遍历待切分的中文句子,根据词典中的词语进行匹配。如果匹配成功,则将匹配的词语作为一个词切分出来。如果没有匹配成功,则按字符切分。最后,返回切分结果。
max_backward_cut方法实现了最大向后匹配算法。它从右向左遍历待切分的中文句子,根据词典中的词语进行匹配。如果匹配成功,则将匹配的词语作为一个词切分出来。如果没有匹配成功,则按字符切分。最后,将切分结果倒序返回。
max_biward_cut方法实现了双向最大匹配算法。它先调用max_forward_cut方法和max_backward_cut方法分别得到正向和逆向的切分结果。然后,根据启发式规则比较两种切分结果,决定正确的分词方法。如果正向和逆向的切分结果词数不同,则选择词数较少的那个。如果词数相同,则根据单字数量选择切分结果。
双向最大匹配法是将正向最大匹配法得到的分词结果和逆向最大匹配法得到的结果进行比较,从而决定正确的分词方法。
启发式规则:
1.如果正反向分词结果词数不同,则取分词数量较少那个;
2.如果分词结果词数相同:
a.分词结果相同,说明没有歧义,可以返回任意一个结果;
b.分词结果不同,返回其中单字较少的那个。
在最大向前匹配和最大向后匹配中,算法会从待切分的汉语句的左侧或右侧开始,每次选择当前位置开始的最长匹配字段,然后移动到下一个未匹配的位置。这个过程一直进行,直到整个句子被切分完毕。在双向最大向前匹配中,先使用最大向前匹配和最大向后匹配分别得到两组分词结果,然后根据一定的启发式规则来确定最终的分词结果。这种方法可以一定程度上解决中文分词中的歧义问题。
总体来说,最大匹配法是一种贪心算法,它每次选择当前位置的最长匹配字段,希望通过局部最优的选择达到整体的最优结果。
然后我们运行build_data.py脚本,通过collect_medical函数完成数据的收集整理工作。
再将整理好的数据重新存入数据库中。在init文件里配置数据库连接,找到当前文件所在的目录,指定连接的数据库及其下面的collection。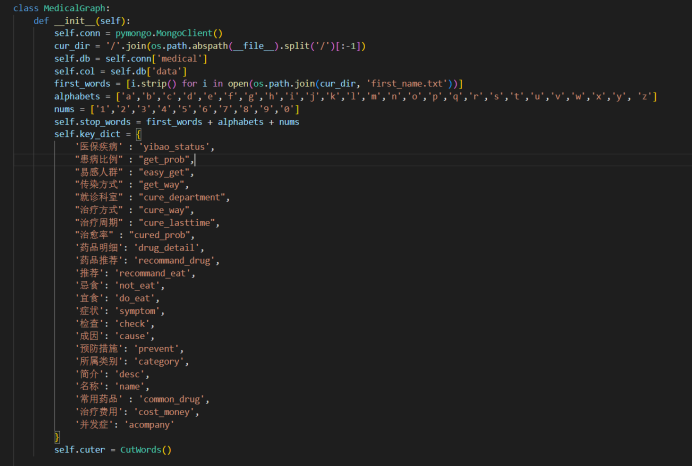
再利用python脚本导出爬出的医疗数据并设置格式为data.json格式文件:
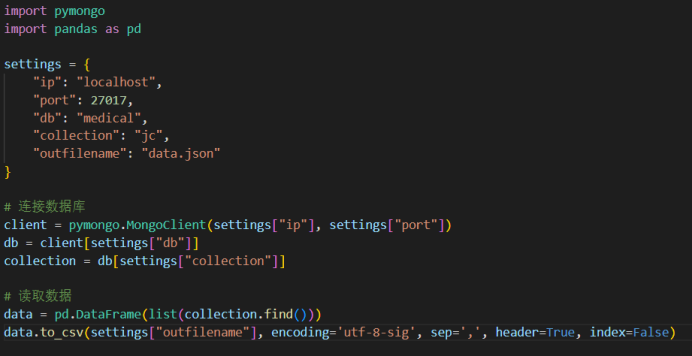

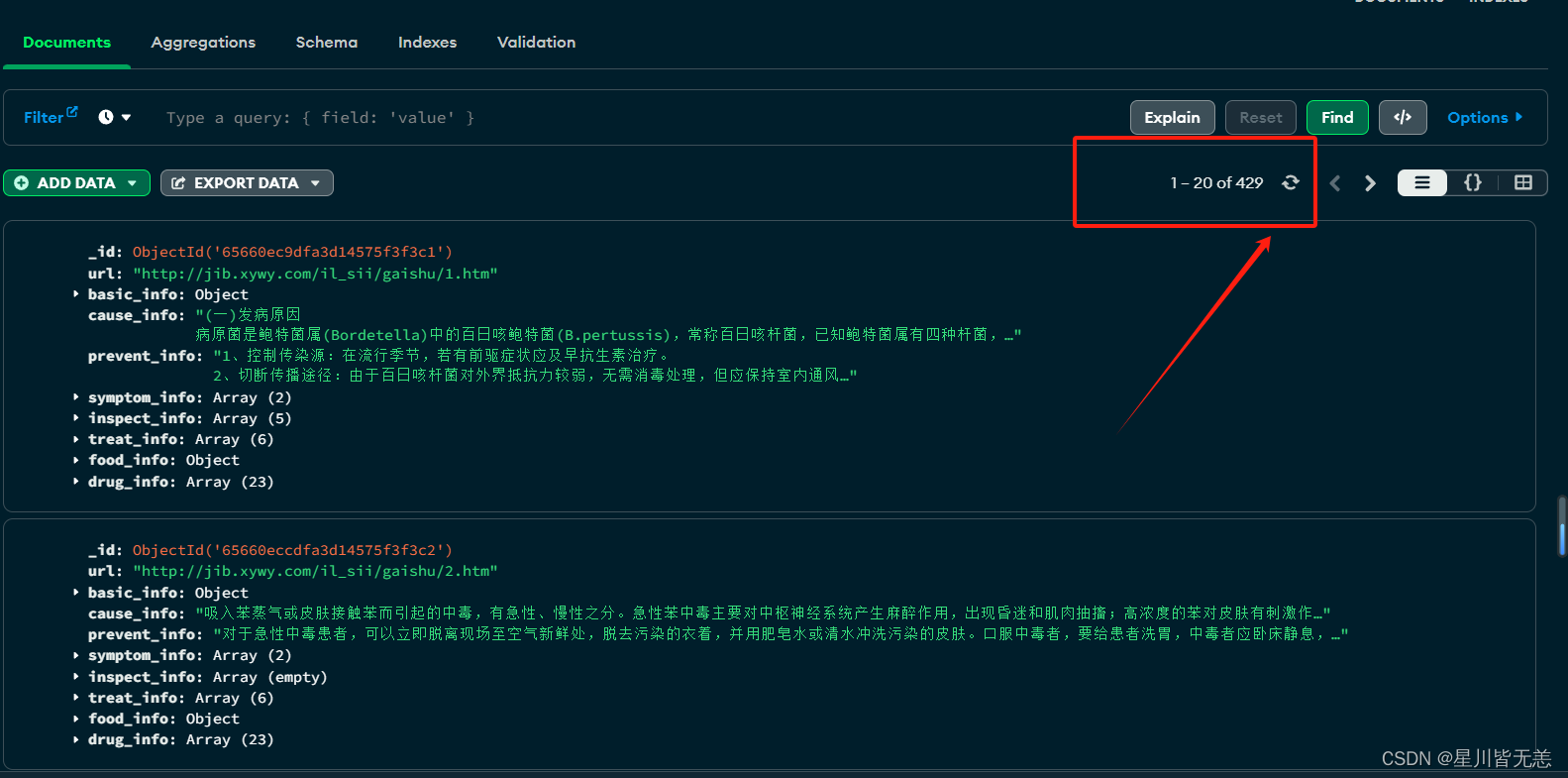
处理数据后对应的图谱系统数据关键词:
将从MongoDB导出的data.json数据格式处理之后保存为medical.json和medical2.json文件,其中medical2.json为medical.json中的部分数据。
考虑到构建全部知识图谱需要很久,所以取出一部分数据(medical2.json)先进行构建图谱。
处理后结构化的数据保存到excel文件:
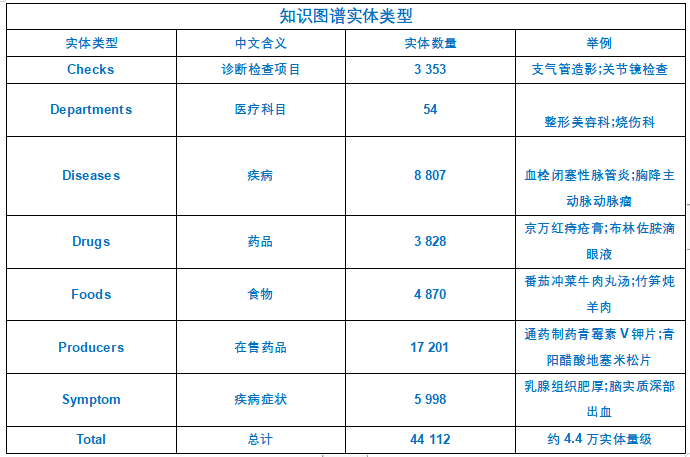
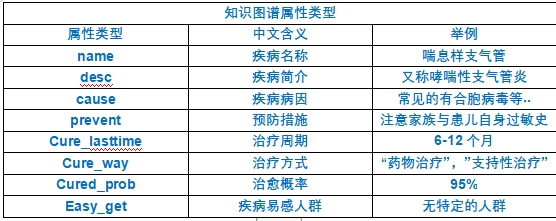
3)所搭建的系统框架,包括知识图谱实体类型,实体关系类型,知识图谱属性类型等。
知识图谱实体类型
一般来说,一个医疗知识图谱问答系统Schema包括以下几个部分:
实体:指代医疗领域中的具体概念或对象,如药品、疾病、症状等。
属性:指代实体的特征或描述,如药品的成分、剂型、适应症等。
关系:指代实体之间的联系或影响,如疾病与药物的治疗关系、食物的忌吃关系等。
问题:指代用户对医疗领域的信息需求,如“高血压应该吃什么药?”、“感冒有哪些常见的症状?”等。
答案:指代针对问题的回复或解释,如“高血压可以服用降压药物,如氨氯地平片、硝苯地平片等。”、“感冒常见的症状有发热、咳嗽、流鼻涕等。”等。
departments = [] #科室 diseases = [] # 疾病 drugs = [] # 药品 foods = [] # 食物 producers = [] #药品大类 symptoms = []#症状 
实体关系类型
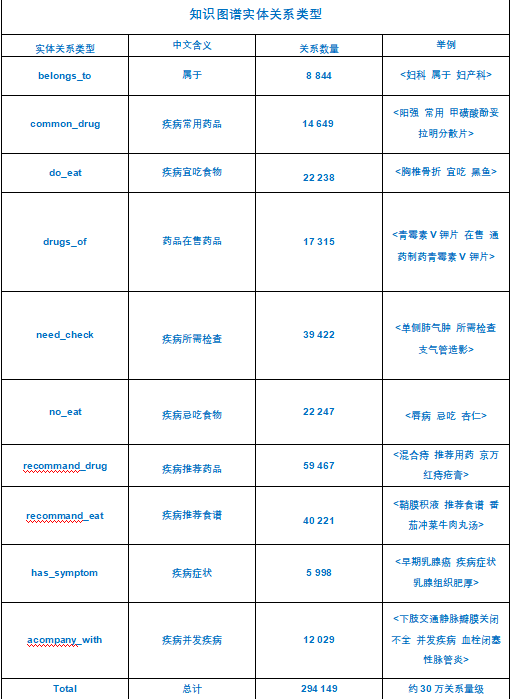
# 构建节点实体关系,共11类,medical2做出来的只有10类,因为数据量少 rels_department = [] rels_noteat = [] # 疾病-忌吃食物关系 rels_doeat = [] # 疾病-宜吃食物关系 rels_recommandeat = [] # 疾病-推荐吃食物关系 rels_commonddrug = [] # 疾病-通用药品关系 rels_recommanddrug = [] # 疾病-热门药品关系 rels_check = [] # 疾病-检查关系 rels_drug_producer = [] # 厂商-药物关系 rels_symptom = [] #疾病症状关系 rels_acompany = [] # 疾病并发关系 rels_category = [] # 疾病与科室之间的关系 知识图谱属性类型
医疗问答KBQA查询功能表
4)导入Neo4j数据库,生成图谱。
新建一个数据库:基于医疗领域的问答系统
六、 构建neo4j知识图谱
开启Node4j数据库:
连接数据库 通过bolt协议(数据传输更快)
对绝对路径进行拼接 获取json文件路径 这里使用的Json为部分数据量,这里数据txt和json格式都有,我们用json就可以了。
连接我们所建的neo4j数据库:
知识图谱建模
在数据库中构建了三元组并且这些三元组形成了结构化的数据关系,进行知识图谱建模。知识图谱是一种用于表示和组织实体、属性和它们之间关系的图形结构。三元组(Subject, Predicate, Object)是知识图谱中的基本构建块,每个三元组都可以被看作是一个图中的边,连接两个节点(实体)。这些节点和边的组合形成了一个图,其中节点表示实体,边表示实体之间的关系。这种方式可以用于构建一个基本的知识图谱。
具体以下几个方面:
实体(Entities): 你的数据库中的记录是知识图谱中的实体。每个实体对应于一个节点。
属性(Properties): 数据库中的字段可以被视为实体的属性。这些属性可以形成连接实体的边。
关系(Relationships): 三元组中的谓词(Predicate)对应于实体之间的关系。关系连接两个实体。
本体(Ontology): 如果你想要对实体和关系进行更详细的定义和注释,可以考虑使用本体。
实体识别和定义
节点类型的定义:
checks, departments, diseases, drugs, foods, producers, symptoms: 这些都是不同类型的节点,代表了医学领域中的检查、科室、疾病、药品、食物、药品大类和症状等实体。
实体属性的定义:
name: 疾病的名称。
desc: 描述疾病的属性。
prevent: 预防措施。
cause: 引起疾病的原因。
easy_get: 疾病容易发生的原因或情况。
cure_department: 治疗疾病的科室。
cure_way: 治疗方式。
cure_lasttime: 治疗持续时间。
symptom: 疾病的症状。
cured_prob: 治愈概率。
关系抽取和定义
关系抽取
症状关系抽取
pythonif 'symptom' in data_json: symptoms += data_json['symptom'] *# 组合症状列表* for symptom in data_json['symptom']: rels_symptom.append([disease, symptom]) *# 对于每个症状建立一个疾病——症状的关系* \1. 如果数据中包含了关于症状的信息('symptom’字段),则将这些症状添加到症状列表中,并为每个症状建立一个与当前疾病相关的关系。
并发症关系抽取
pythonif 'acompany' in data_json: for acompany in data_json['acompany']: rels_acompany.append([disease, acompany]) *# 建立一个疾病——伴随疾病的关系* \2. 如果数据中包含了关于并发症的信息('acompany’字段),则为每个并发症建立一个与当前疾病相关的关系。
其他属性关系抽取
pythonif 'desc' in data_json: disease_dict['desc'] = data_json['desc'] *# 定义疾病描述为属性而非关系* *# 类似地处理预防、原因、发病率、易感人群等属性* \3. 对于疾病的描述、预防方法、引起原因、发病率、易感人群等信息,将其作为疾病节点的属性而非建立关系。
治疗科室关系抽取
pythonif 'cure_department' in data_json: cure_department = data_json['cure_department'] **if** len(cure_department) == 1: rels_category.append([disease, cure_department[0]]) *# 只有一个科室的情况* **if** len(cure_department) == 2: big = cure_department[0] *# 大科室* small = cure_department[1] *# 细分小科室* rels_department.append([small, big]) *# 提取科室——科室关系* rels_category.append([disease, small]) *# 提取疾病——科室关系* disease_dict['cure_department'] = cure_department departments += cure_department \4. 如果数据中包含了关于治疗科室的信息('cure_department’字段),则根据科室的数量,建立疾病与科室之间的关系。如果只有一个科室,建立疾病与该科室的关系;如果有两个科室,建立疾病与细分小科室的关系,同时提取科室之间的关系。
治疗途径关系抽取
pythonif ‘cure_way’ in data_json:
disease_dict[‘cure_way’] = data_json[‘cure_way’] # 治疗途径作为疾病节点的属性
\5. 如果数据中包含了关于治疗途径的信息('cure_way’字段),则将其作为疾病节点的属性而非建立关系。
这段代码主要通过判断JSON数据中是否包含特定字段,从而提取与疾病相关的信息,并构建了疾病知识图谱的关系。关系主要包括症状、并发症、治疗科室等方面,而属性则包括疾病的描述、预防方法、引起原因、发病率、易感人群以及治疗途径等。
if 'cure_lasttime' in data_json: disease_dict['cure_lasttime'] = data_json['cure_lasttime']#治疗时间 if 'cured_prob' in data_json: disease_dict['cured_prob'] = data_json['cured_prob']#治愈概率 if 'common_drug' in data_json: common_drug = data_json['common_drug']#常用药物 for drug in common_drug: rels_commonddrug.append([disease, drug])#提取疾病——药物的关系 drugs += common_drug if 'recommand_drug' in data_json: recommand_drug = data_json['recommand_drug']#推荐药物 drugs += recommand_drug for drug in recommand_drug: rels_recommanddrug.append([disease, drug])#提取疾病——推荐药物的关系 if 'not_eat' in data_json: not_eat = data_json['not_eat']#不能吃的食物 for _not in not_eat: rels_noteat.append([disease, _not])#提取疾病——不能吃的食物的关系 foods += not_eat do_eat = data_json['do_eat']#可以吃的食物 for _do in do_eat: rels_doeat.append([disease, _do])#提取疾病——能吃的食物的关系 foods += do_eat recommand_eat = data_json['recommand_eat']#推荐吃的食物 for _recommand in recommand_eat: rels_recommandeat.append([disease, _recommand])#提取疾病——推荐吃的食物的关系 foods += recommand_eat if 'check' in data_json: check = data_json['check']#检查项,一个疾病对应多个检查 for _check in check: rels_check.append([disease, _check])#提取疾病——检查项的关系 checks += check if 'drug_detail' in data_json: drug_detail = data_json['drug_detail']#药物详细信息 producer = [i.split('(')[0] for i in drug_detail] rels_drug_producer += [[i.split('(')[0], i.split('(')[-1].replace(')', '')] for i in drug_detail] producers += producer disease_infos.append(disease_dict)#添加疾病信息list 上面代码段主要涉及疾病相关的信息和与之相关的关系抽取。下面是对每个条件的详细分析:
*治疗时间和治愈概率关系抽取*:
\6. 如果 cure_lasttime 在 data_json 中,将其值存储到 disease_dict[‘cure_lasttime’] 中,表示疾病的治疗时间。
\7. 如果 cured_prob 在 data_json 中,将其值存储到 disease_dict[‘cured_prob’] 中,表示疾病的治愈概率。
*常用药物和推荐药物关系抽取*:
\8. 如果 common_drug 在 data_json 中,遍历每个药物,将疾病和药物的关系存储在 rels_commonddrug 列表中,并将药物添加到 drugs 中。
\9. 如果 recommand_drug 在 data_json 中,遍历每个药物,将疾病和药物的关系存储在 rels_recommanddrug 列表中,并将药物添加到 drugs 中。
*食物与疾病关系抽取*:
\10. 如果 not_eat 在 data_json 中,遍历不能吃的食物,将疾病和食物的关系存储在 rels_noteat 列表中,并将食物添加到 foods 中。
\11. 如果 do_eat 在 data_json 中,遍历可以吃的食物,将疾病和食物的关系存储在 rels_doeat 列表中,并将食物添加到 foods 中。
\12. 如果 recommand_eat 在 data_json 中,遍历推荐吃的食物,将疾病和食物的关系存储在 rels_recommandeat 列表中,并将食物添加到 foods 中。
*检查项与疾病关系抽取*:
\13. 如果 check 在 data_json 中,遍历每个检查项,将疾病和检查项的关系存储在 rels_check 列表中,并将检查项添加到 checks 中。
*药物详细信息中的生产商关系抽取*:
\14. 如果 drug_detail 在 data_json 中,通过对每个药物详细信息的处理,提取生产商信息,将疾病和生产商的关系存储在 rels_drug_producer 列表中,并将生产商信息添加到 producers 中。
return set(drugs), set(foods), set(checks), set(departments), set(producers), set(symptoms), set(diseases), disease_infos,\ rels_check, rels_recommandeat, rels_noteat, rels_doeat, rels_department, rels_commonddrug, rels_drug_producer, rels_recommanddrug,\ rels_symptom, rels_acompany, rels_category 使用了 Python 中的 return 语句,返回了多个集合和列表。这些变量包括:
\1. set(drugs): 药物的集合
\2. set(foods): 食物的集合
\3. set(checks): 检查项的集合
\4. set(departments): 科室的集合
\5. set(producers): 生产商的集合
\6. set(symptoms): 症状的集合
\7. set(diseases): 疾病的集合
\8. disease_infos: 包含疾病信息的列表
\9. rels_check: 检查项关系的列表
\10. rels_recommandeat: 推荐食物关系的列表
\11. rels_noteat: 不能吃食物关系的列表
\12. rels_doeat: 可以吃食物关系的列表
\13. rels_department: 科室关系的列表
\14. rels_commonddrug: 常用药物关系的列表
\15. rels_drug_producer: 药物和生产商关系的列表
\16. rels_recommanddrug: 推荐药物关系的列表
\17. rels_symptom: 症状关系的列表
\18. rels_acompany: 伴随症状关系的列表
\19. rels_category: 类别关系的列表
每个集合或列表包含了相应类型的信息,例如药物、食物、症状、疾病等等,并且存在着关系的提取,比如药物和生产商之间的关系、症状和疾病之间的关系等。这些信息和关系的提取有助于对医疗数据进行分析、关联和进一步的应用。这些信息的提取有助于构建一个更全面的医疗知识图谱,为后续的问答系统提供更丰富的信息支持
关系类型定义
rels_department: 科室之间的关系。
rels_noteat, rels_doeat, rels_recommandeat: 疾病与食物之间的关系,包括忌吃、宜吃和推荐吃。
rels_commonddrug, rels_recommanddrug: 疾病与药品之间的关系,包括通用药品和热门药品。
rels_check: 疾病与检查之间的关系。
rels_drug_producer: 药品与厂商之间的关系。
rels_symptom: 疾病与症状之间的关系。
rels_acompany: 疾病与并发症之间的关系。
rels_category: 疾病与科室之间的关系。
'''读取文件''' def read_nodes(self): \# 共7类节点,节点的设置与业务相关 checks = [] # 检查 departments = [] #科室 diseases = [] # 疾病 drugs = [] # 药品 foods = [] # 食物 producers = [] #药品大类 symptoms = []#症状 \#这一项没有出现在节点中,在后面编程中用于创建疾病信息表 disease_infos = []#疾病信息 \# 构建节点实体关系,共11类,medical2做出来的只有10类,因为数据量少 rels_department = [] # 科室-科室关系,这一项关系PPT中未列出,后加入 rels_noteat = [] # 疾病-忌吃食物关系 rels_doeat = [] # 疾病-宜吃食物关系 rels_recommandeat = [] # 疾病-推荐吃食物关系 rels_commonddrug = [] # 疾病-通用药品关系 rels_recommanddrug = [] # 疾病-热门药品关系 rels_check = [] # 疾病-检查关系 rels_drug_producer = [] # 厂商-药物关系 rels_symptom = [] #疾病症状关系 rels_acompany = [] # 疾病并发关系 rels_category = [] # 疾病与科室之间的关系 
七、知识图谱数据入库
根据字典形式的数据创建结点,以疾病为中心定义关系形成三元组表示的知识,将结点和关系导入neo4j数据库形成知识图谱,通过运行build_medicalgraph.py脚本构建图谱:
建立实体关系类型:
该脚本构建了一个MedicalGraph类,定义了Graph类的成员变量g和json数据路径成员变量data_path。
建立的图谱实体关系和属性类型数量有点多,需要等待一会。耐心等待就可以,因为我们这个爬取的数据量大,知识图谱如果数据量不足,构建的实体关系和属性类型再多再合适也没用,大数据下的知识图谱才有更强的问答能力。
建立的图谱实体关系和属性类型数量有点多,需要等待一会。
脚本运行完之后查看neo4j数据库中构建的知识图谱:
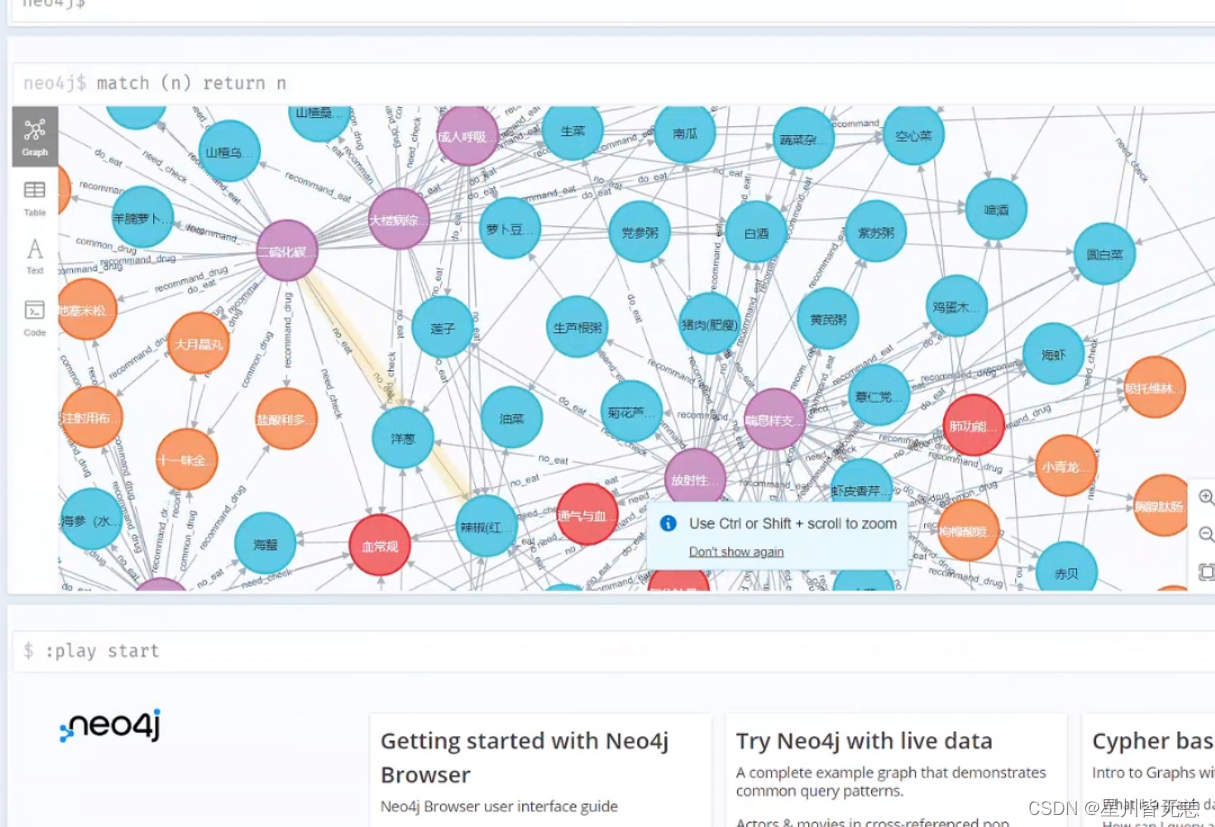
match (n) return n 
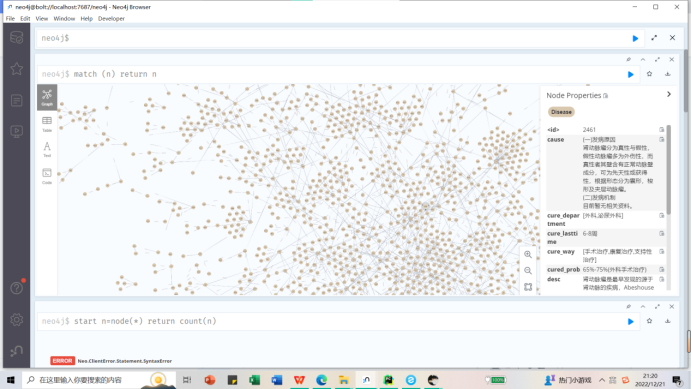
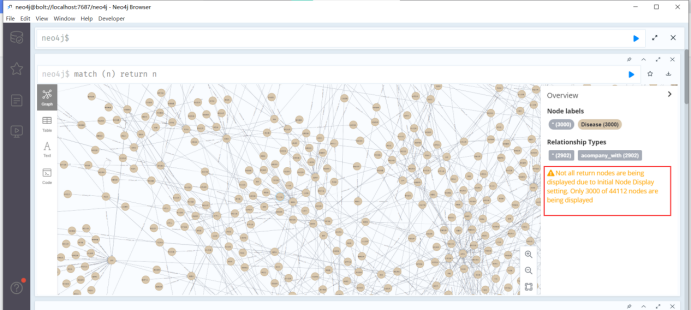
提示:Not all return nodes are being displayed due to Initial Node Display setting. Only 3000 of 44112 nodes are being displayed
由于“初始节点显示”设置,并非所有返回节点都显示。44112个节点中仅显示3000个
这里因为我设置的参数只显示前3000个,4万多个节点只显示了一部分。
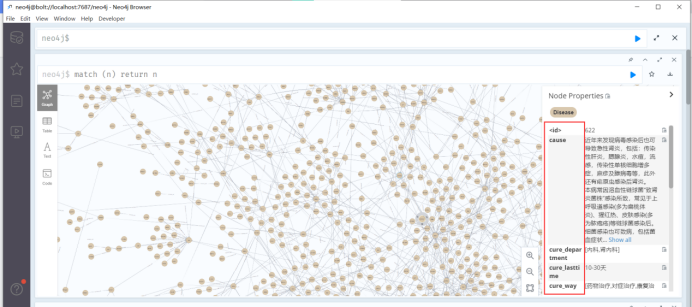
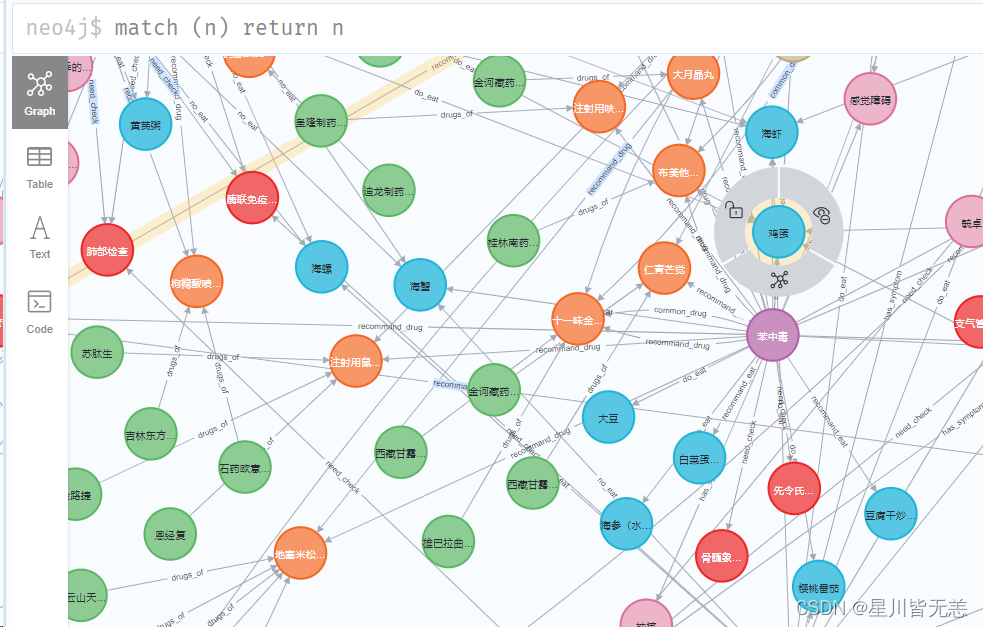
对于单个节点分析是先建一个label为disease、name为疾病名称的node,property方面包括prevent、cure_way、cause等等,另外,对于症状、科室、检查方法等信息都建立单独的node,同时通过has_symptom、belongs_to、need_check等关联把它们和疾病名称关联起来,因为疾病之间存在并发关系,疾病之间也可以通过症状串联起来,所以最后我们利用大量的
医疗数据,就能构建一个大型的医疗知识图谱:
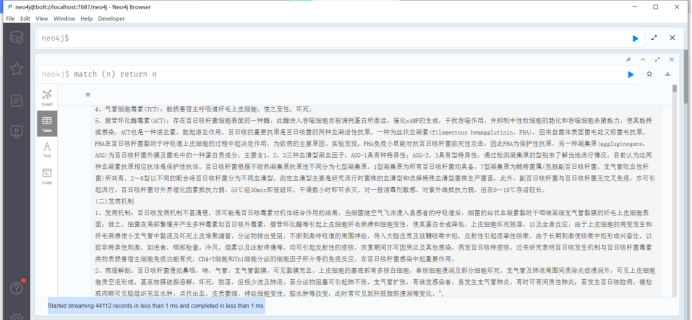
提示:Started streaming 44112 records in less than 1 ms and completed in less than 1 ms.
在不到1毫秒内开始流式传输44112条记录,并在不到1秒内完成。
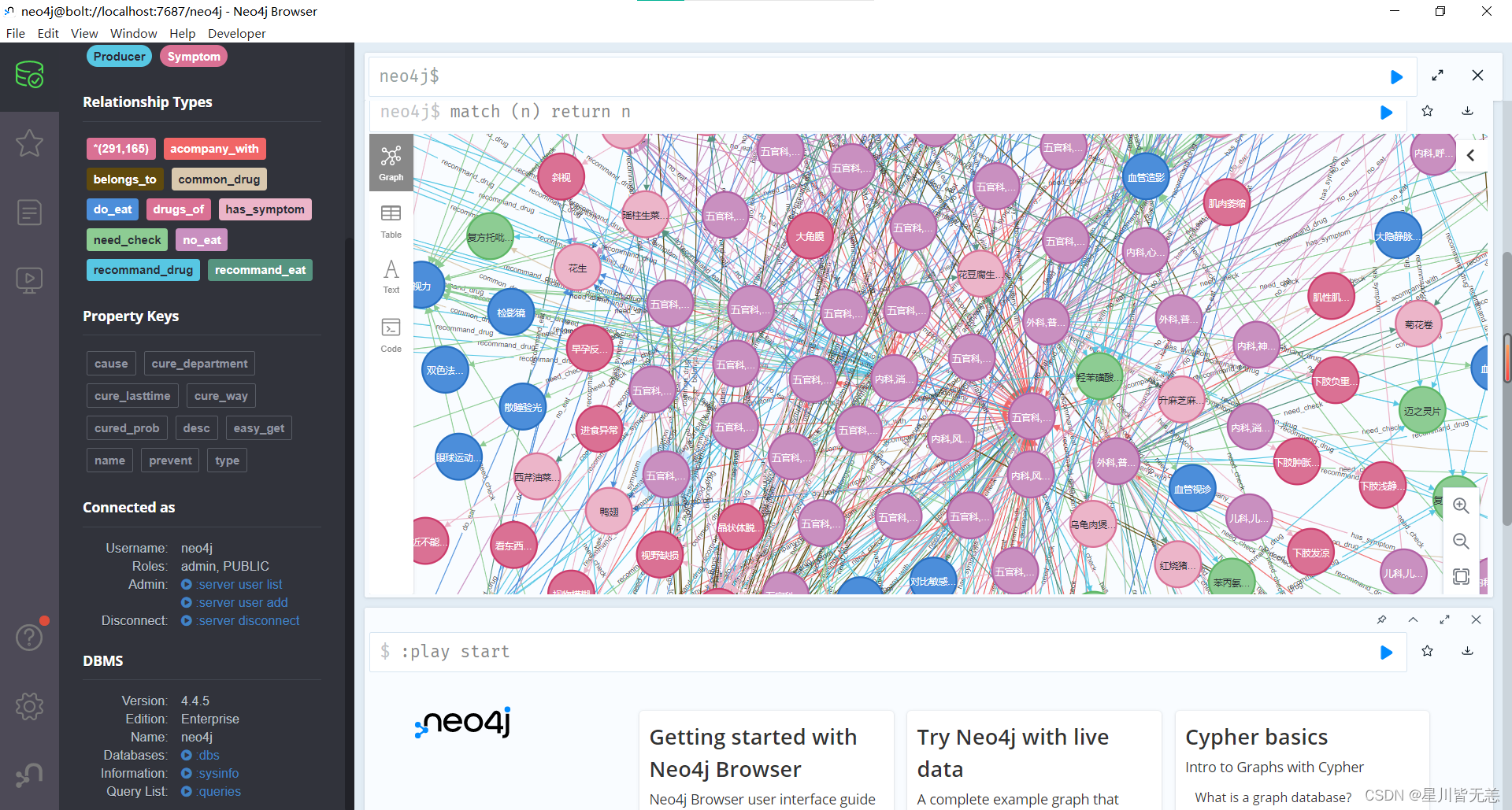
这个节点和类型颜色可以随意更改,选择自己喜欢的颜色就行。
5)Python 与Neo4j的交互实现。
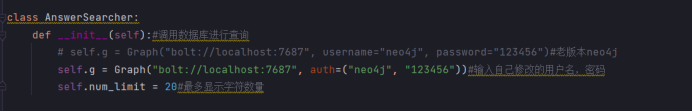
连接Node4j数据库:
from py2neo import Graph class AnswerSearcher: def __init__(self):#调用数据库进行查询 # self.g = Graph("bolt://localhost:7687", username="neo4j", password="123456")#老版本neo4j self.g = Graph("bolt://localhost:7687", auth=("neo4j", "123456"))#输入自己修改的用户名,mima self.num_limit = 20#最多显示字符数量 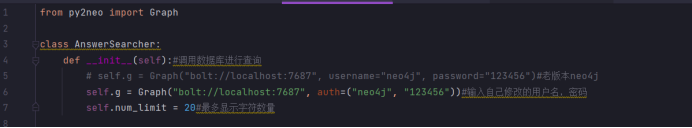
创建图对象:
class MedicalGraph: def __init__(self): cur_dir = '/'.join(os.path.abspath(__file__).split('/')[:-1]) self.data_path = os.path.join(cur_dir, 'data/medical.json') # self.g = Graph("http://localhost:7474", username="neo4j", password="123456") self.g = Graph("bolt://localhost:7687", auth=("neo4j", "123456")) 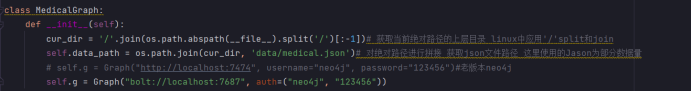
增加node节点:
def create_node(self, label, nodes): count = 0 for node_name in nodes: node = Node(label, name=node_name) self.g.create(node) count += 1 print(count, len(nodes)) return 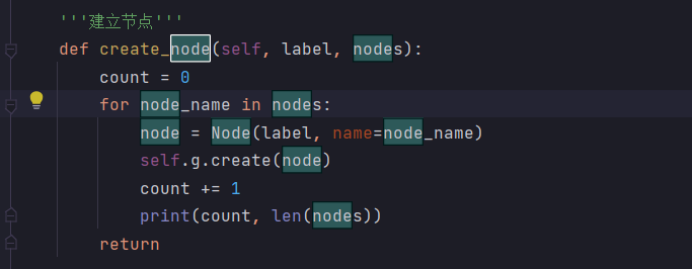
创建实体关系:
def create_graphrels(self): Drugs, Foods, Checks, Departments, Producers, Symptoms, Diseases, disease_infos, rels_check, rels_recommandeat, rels_noteat, rels_doeat, rels_department, rels_commonddrug, rels_drug_producer, rels_recommanddrug,rels_symptom, rels_acompany, rels_category = self.read_nodes() self.create_relationship('Disease', 'Food', rels_recommandeat, 'recommand_eat', '推荐食谱')#调用下面的关系边创建函数 self.create_relationship('Disease', 'Food', rels_noteat, 'no_eat', '忌吃') self.create_relationship('Disease', 'Food', rels_doeat, 'do_eat', '宜吃') self.create_relationship('Department', 'Department', rels_department, 'belongs_to', '属于') self.create_relationship('Disease', 'Drug', rels_commonddrug, 'common_drug', '常用药品') self.create_relationship('Producer', 'Drug', rels_drug_producer, 'drugs_of', '生产药品') self.create_relationship('Disease', 'Drug', rels_recommanddrug, 'recommand_drug', '好评药品') self.create_relationship('Disease', 'Check', rels_check, 'need_check', '诊断检查') self.create_relationship('Disease', 'Symptom', rels_symptom, 'has_symptom', '症状') self.create_relationship('Disease', 'Disease', rels_acompany, 'acompany_with', '并发症') self.create_relationship('Disease', 'Department', rels_category, 'belongs_to', '所属科室') 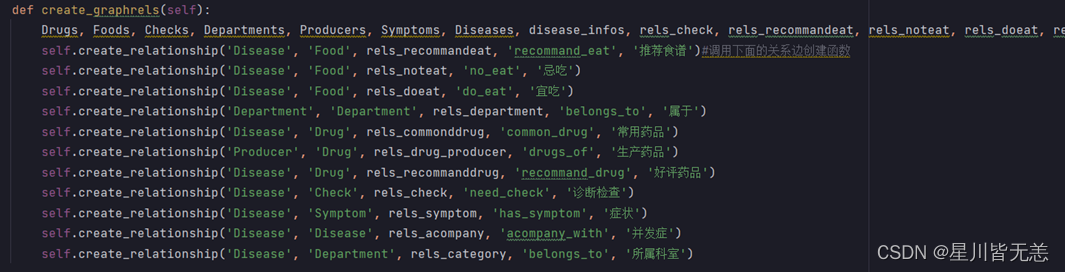
更新节点:
更新先要找出Nodes,再使用事务的push更新
from py2neo import Graph, Node, Relationship, NodeMatcher, Subgraph g = Graph('http://localhost:7687', auth=("neo4j", "123456")) from py2neo import Graph, NodeMatcher tx = g.begin()# 找到要找的Nodes matcher = NodeMatcher(g) init_node = matcher.match("Person", name="节点名称") new_node = init_node.first() new_node['name'] = "新的节点名称" sub = Subgraph(nodes=[new_node]) tx.push(sub) g.commit(tx) 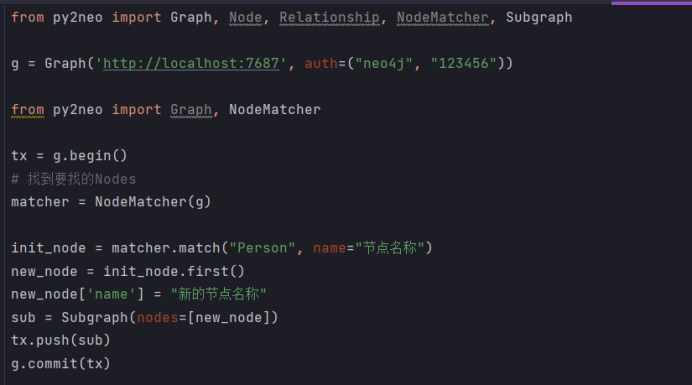
删除关系链(节点也会删除):
from py2neo import Graph, Node, Relationship, NodeMatcher, Subgraph, RelationshipMatcher g = Graph('http://localhost:7687', auth=("neo4j", "123456")) matcher = NodeMatcher(g) r_matcher = RelationshipMatcher(g) fugui = matcher.match('Person', name='name1').first() youqian = matcher.match('Person', name='name2').first() relation = r_matcher.match(nodes=[fugui, youqian]).first() #也可以是none,表示任意节点 print(relation) g.delete(relation) 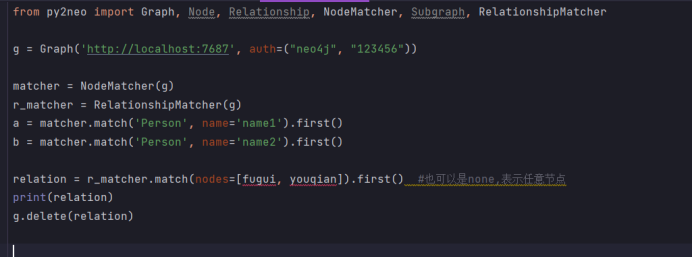
两个节点新加关系:
from py2neo import Graph, Node, Relationship, NodeMatcher, Subgraph g = Graph('http://localhost:7687', auth=("neo4j", "123456")) matcher = NodeMatcher(g) fugui = matcher.match('Person', name='节点名称1').first() youqian = matcher.match('Person', name='节点名称2').first() relation = Relationship(节点名称1, '所要建立的关系', 节点名称2) g.create(relation) 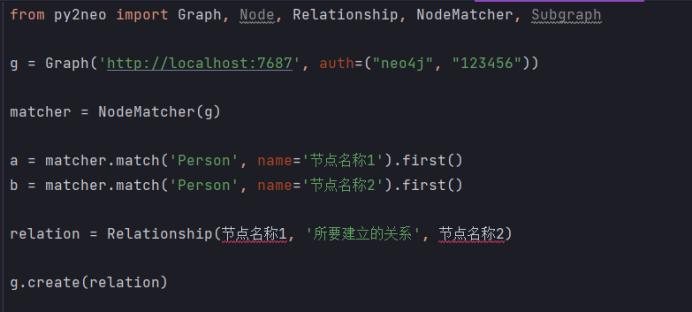
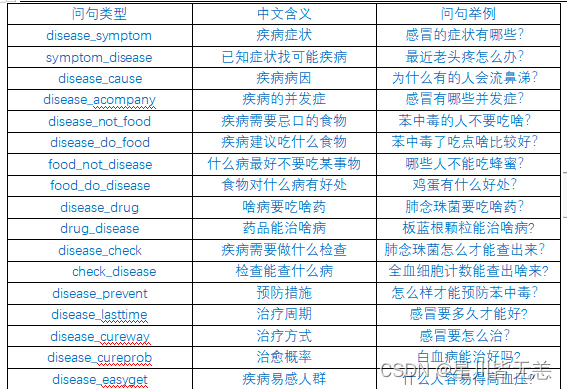
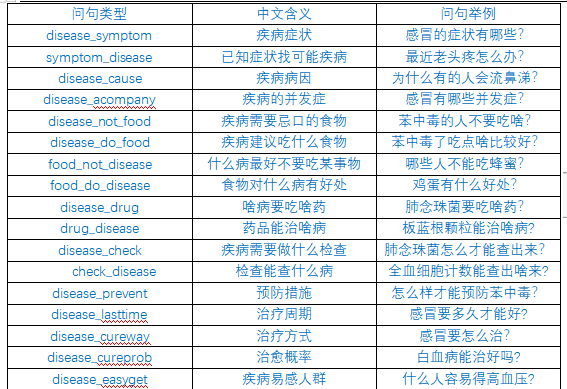
问答系统支持的问答类型
本项目问答对话系统的分析思路,整体上接近一个基于规则的对话系统,首先我们需要对用户输入进行分类,其实就是分析用户输入涉及到的实体及问题类型,也就是Neo4j中的node、property、relationship,然后我们利用分析出的信息,转化成Neo4j的查询语句,最后再把查询的结果返回给用户,就完成了一次问答。
本项目问答系统支持的问答类型:
问答系统整体上涉及到三个模块,问题的分类、问题的解析以及回答的搜索。
对于property和relationship,我们先预设定一系列的问句疑问词,从而对问句的每个词进行对比分析,判断出问句的类型
Aho-Corasick算法
这里需要用到Python的Ahocorasick库进行模式匹配,因此记录该库所使用的到的Aho-Corasick算法(AC算法),Aho-Corasick算法是多模式匹配中的经典算法,目前在实际应用中较多。Aho-Corasick算法对应的数据结构是Aho-Corasick自动机,简称AC自动机Automaton。该算法能够识别出一个给定的语句中包含了哪些词典库中特定的词语,具有很有的模式匹配作用。
算法主要分为以下三个部分:
- 构造Goto表:成功转移到另一个状态
- 构造Failture指针:如果某状态发生匹配失败,需要跳转到一个特定的节点
- 匹配:匹配成功某一字符串
我们构建一个基于Aho-Corasick算法的trie树,用于加速过滤敏感词汇或关键词。
'''构造actree,加速过滤''' def build_actree(self, wordlist): actree = ahocorasick.Automaton() # 初始化trie树,ahocorasick 库 ac自动化 自动过滤违禁数据 for index, word in enumerate(wordlist): actree.add_word(word, (index, word)) # 向trie树中添加单词 actree.make_automaton() # 将trie树转化为Aho-Corasick自动机 return actree 使用Aho-Corasick自动机的目的是在输入文本中高效地检测和过滤多个关键词。这种数据结构的优势在于,它能够同时匹配多个关键词而无需多次扫描输入文本,因此在过滤大量文本时性能较高。
ahocorasick.Automaton(): 初始化一个Aho-Corasick自动机,使用了ahocorasick库。Aho-Corasick算法是一种多模式匹配算法,用于在输入文本中同时查找多个关键词。for index, word in enumerate(wordlist):: 对给定的关键词列表进行遍历。enumerate函数用于同时获取关键词在列表中的索引和关键词本身。actree.add_word(word, (index, word)): 向Aho-Corasick自动机中添加关键词。这里的关键词是来自于wordlist列表,(index, word)是一个元组,包含关键词在列表中的索引和关键词本身。Aho-Corasick算法将这些关键词构建成一个trie树。actree.make_automaton(): 将trie树转化为Aho-Corasick自动机。这一步是Aho-Corasick算法的关键,它会为每个节点添加失败转移(failure transition)和输出(output)信息,使得在匹配过程中能够快速定位到匹配的关键词。return actree: 返回构建好的Aho-Corasick自动机。加粗样式

然后,根据问句的类型和之前识别出来的实体,基于规则推断出property和relationship: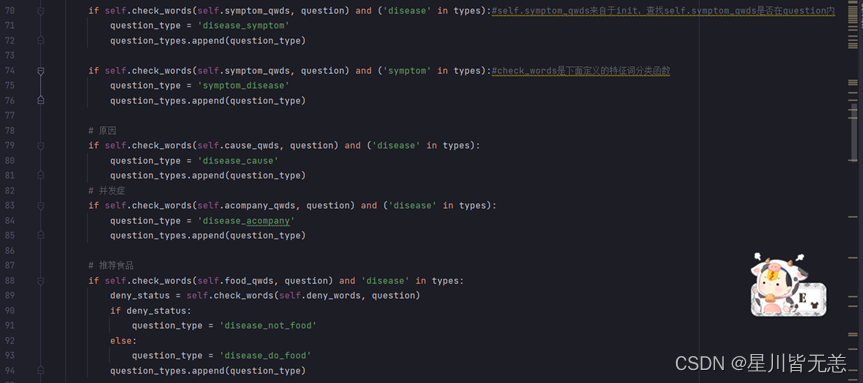
把问题转化为Neo4j的Cypher语句,其实也是预先写好Cypher语句的模板,根据实际的情况把之前分析得到的node、property、relationship填入Cypher语句中进行查询。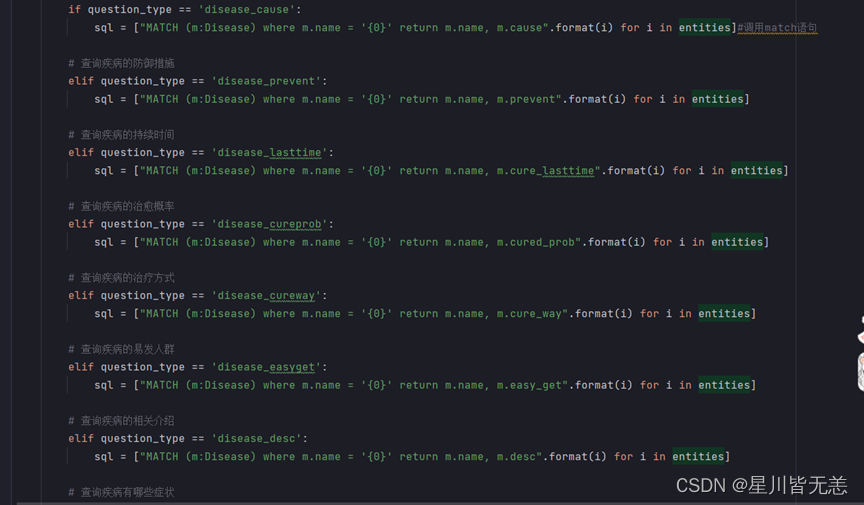
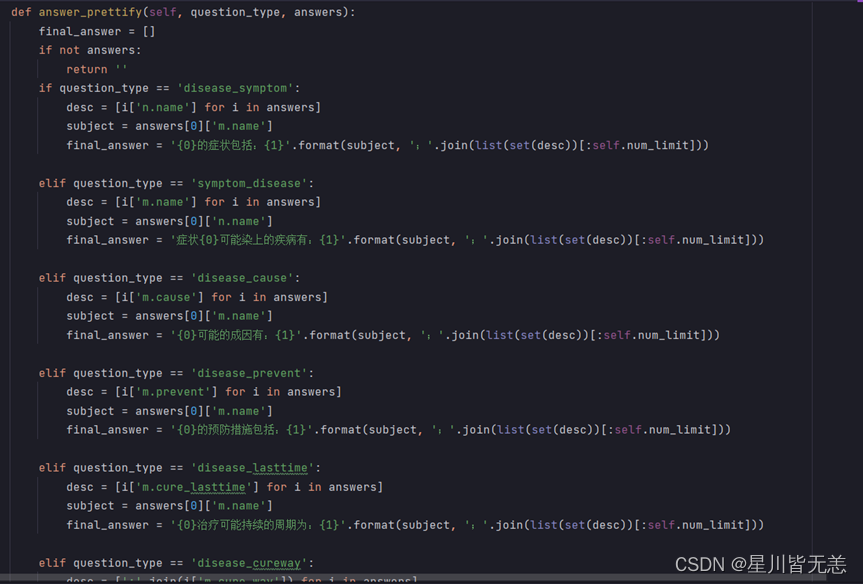
得到了Cypher语句,我们就能连接Neo4j数据库进行查询,得到结果之后,还需要对语句进行一点微调,根据对应的qustion_type,调用相应的回复模板:
八、基于Cypher查询
节点属性查询
使用节点的属性进行匹配。假设节点有一个名为 “name” 的属性,可以按照以下方式修改Cypher查询:
MATCH (n {name: '节点的名字'})-[r]->(m) RETURN n, r, m 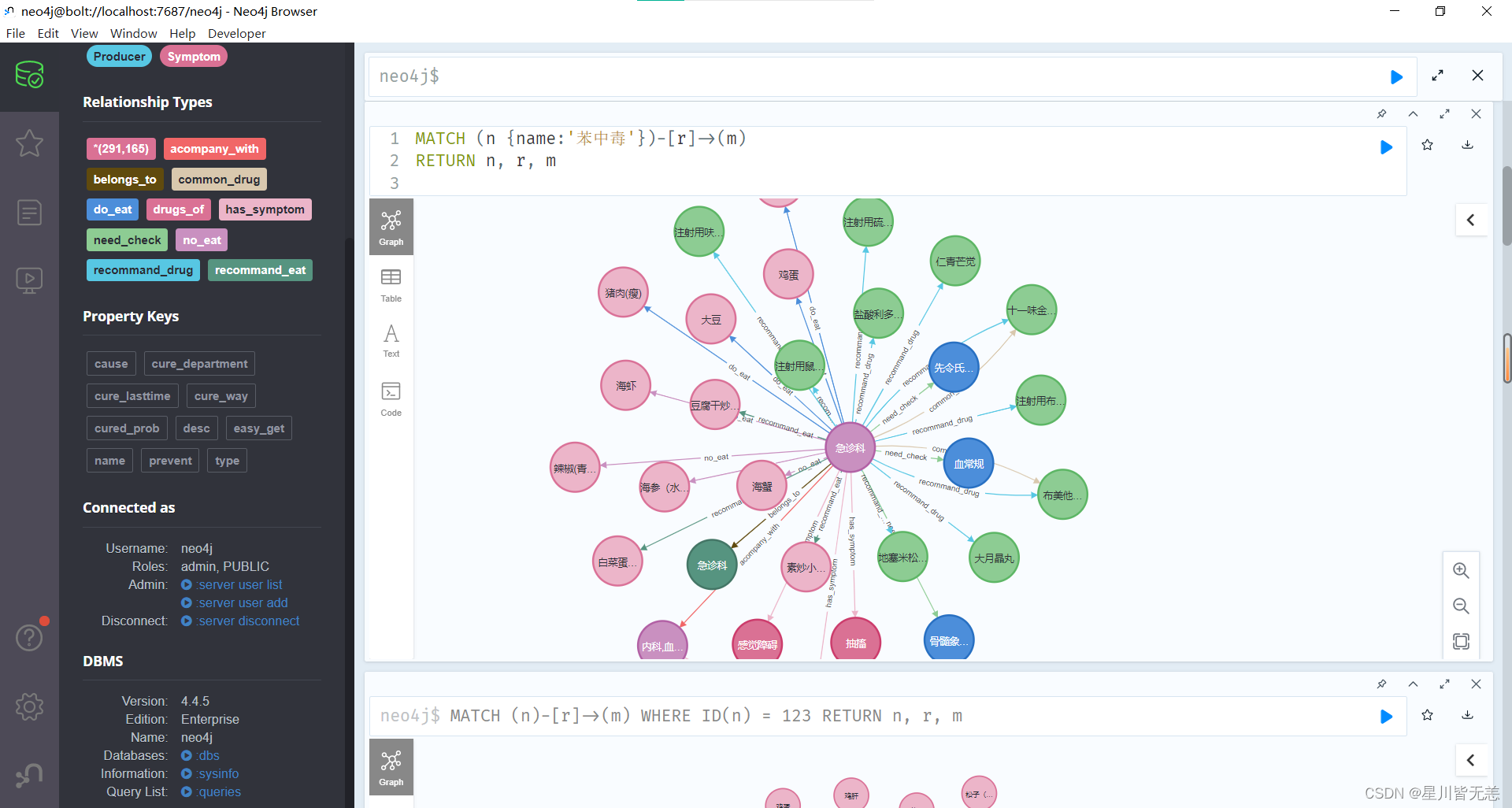
节点ID查询
MATCH (n)-[r]->(m) WHERE ID(n) = 123 RETURN n, r, m 查询匹配 ID 为 123 的节点及其出边的关系和连接的节点。你可以替换 ID(n) = 123 为你实际节点的标识条件。
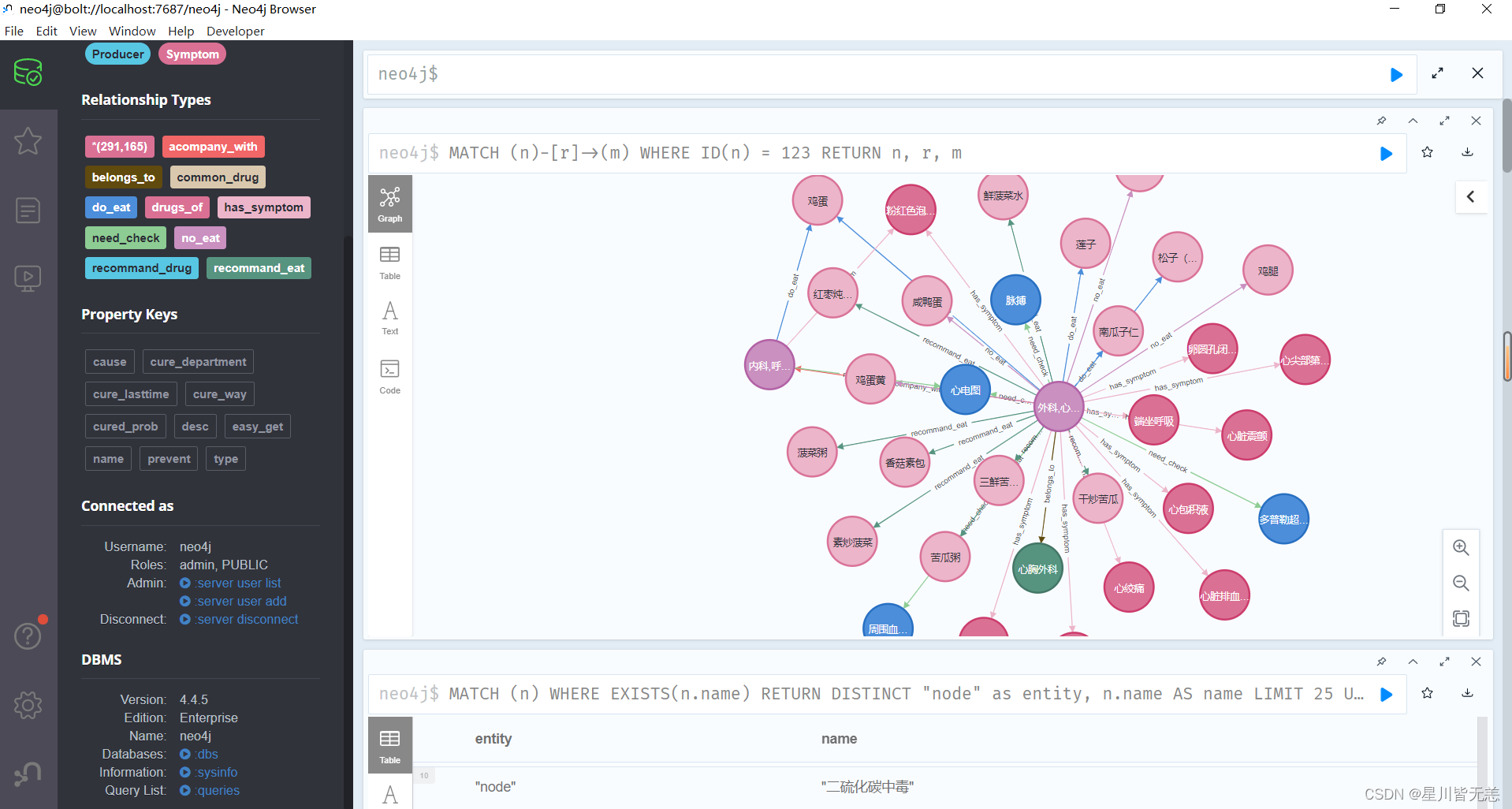
查询所有节点及其关系
MATCH (n)-[r]->(m) RETURN n, r, m 这个查询将返回图中所有节点及其出边的关系和连接的节点。
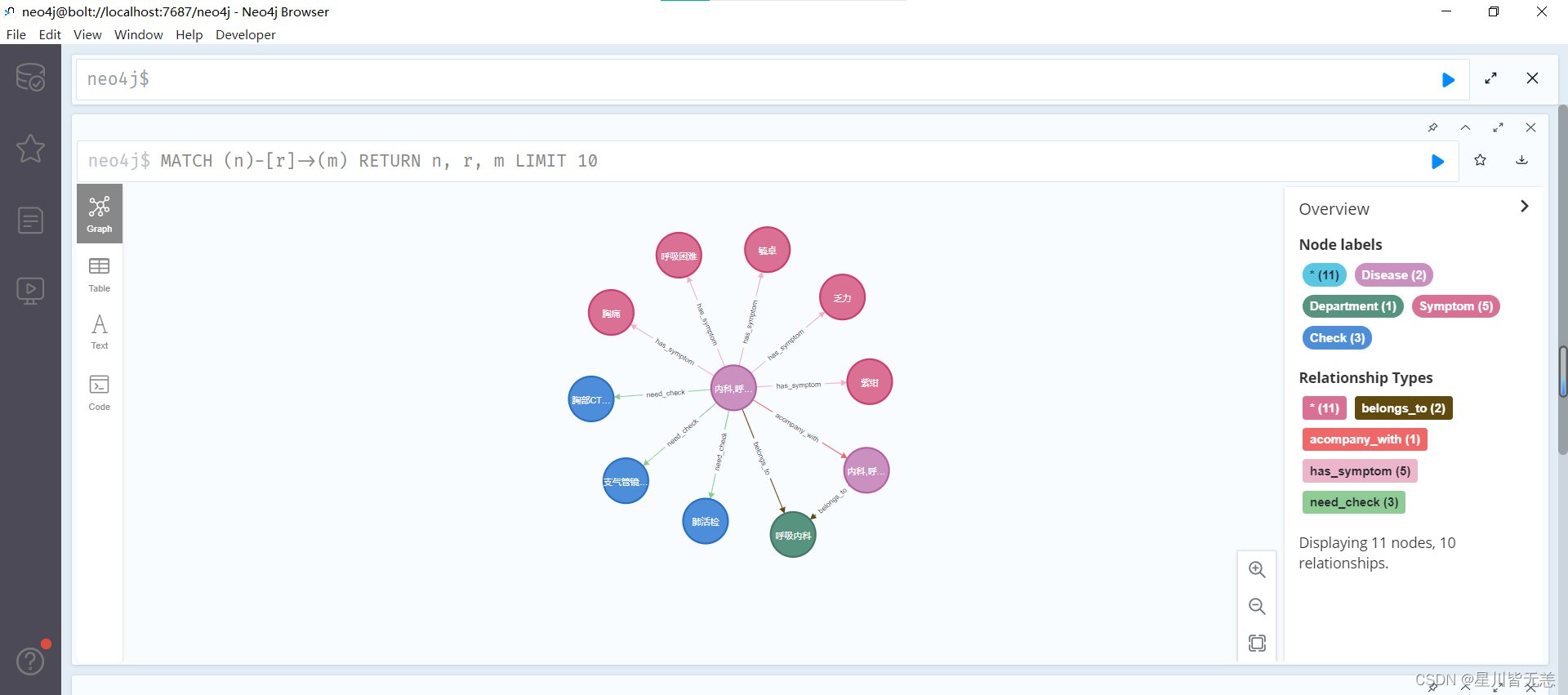
指定关系类型的查询
MATCH (n)-[r:RELATION_TYPE]->(m) RETURN n, r, m 这个查询将匹配指定类型的关系。
限制结果数量
MATCH (n)-[r]->(m) RETURN n, r, m LIMIT 10 这个查询将返回前10个匹配的节点及其关系。
通过节点关系查询
在Neo4j图数据库中查找标签为 acompany_with 的关系,返回前25个匹配的路径。找到所有含有 acompany_with 关系的路径,并返回这些路径的信息,但仅限于前25个结果。这样的查询对于查找与给定关系类型相关的节点和关系的拓扑结构非常有用。
MATCH p=()-[r:acompany_with]->() RETURN p LIMIT 25 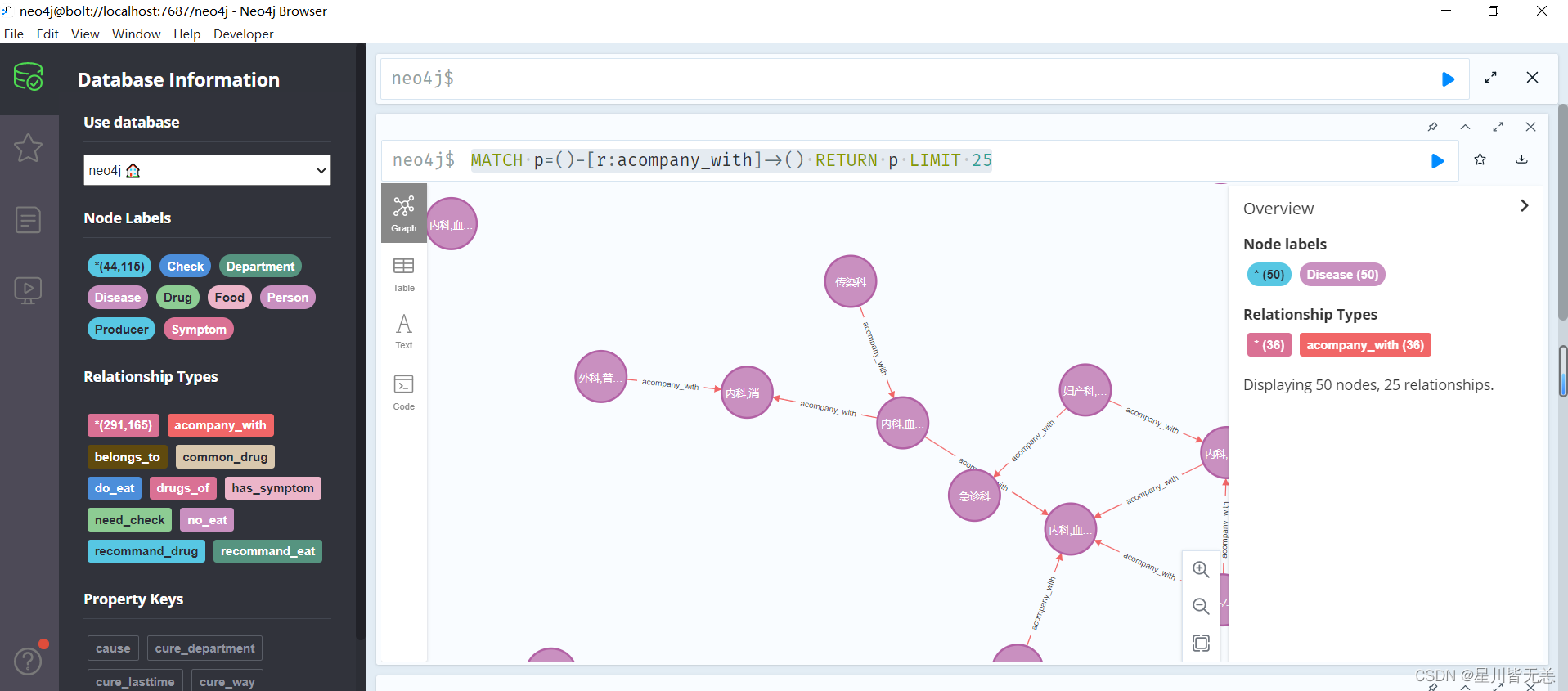
MATCH p=()-[r:acompany_with]->(): 这部分是Cypher的模式匹配语句。它使用MATCH关键字指定了一个模式,其中()-[r:acompany_with]->()描述了一个无向边,该边的关系类型为acompany_with。这个模式中的圆括号表示任意节点,r是关系的别名,acompany_with是关系类型。RETURN p: 这部分指定了在匹配的模式中返回的内容。在这里,p是路径的别名,表示匹配的整个路径(包括节点和关系)。LIMIT 25: 这是一个限制语句,用于指定返回的结果数量,限制为前25个匹配的路径。
这样的查询对于查找与给定关系类型相关的节点和关系的拓扑结构非常有用。
九、项目启动实现方法与步骤
问答框架包含问句分类、问句解析、查询结果三个步骤,具体一步步分析。
首先是问句分类,是通过question_classifier.py脚本实现的。
再通过question_parser.py脚本进行问句分类后对问句进行解析。
然后通过answer_search.py脚本对解析后的结果进行查询
最后通过app.py脚本进行问答实测。
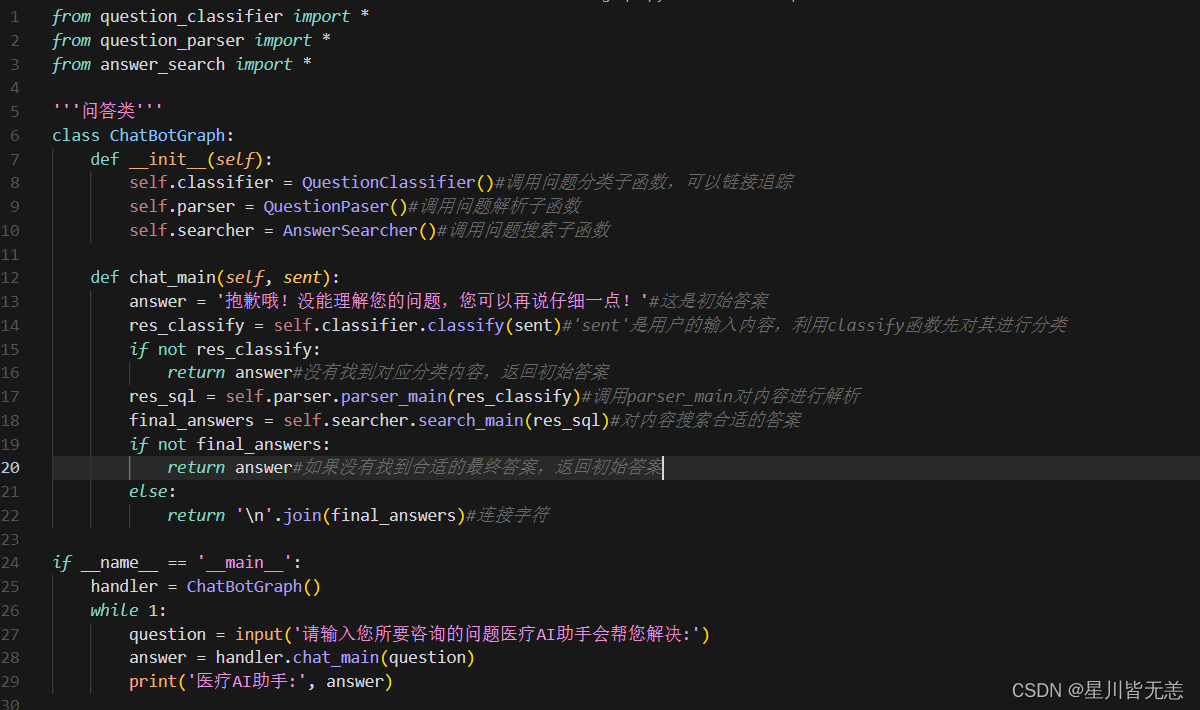
这个chatbot_graph脚本是整个问答系统的主程序。首先创建了一个ChatBotGraph类,其中包含了问题分类器(QuestionClassifier)、问题解析器(QuestionPaser)和答案搜索器(AnswerSearcher)的实例。然后定义了一个chat_main方法,用于处理用户输入的问题。在chat_main方法中,首先将用户输入的问题进行分类,得到分类结果res_classify。如果没有找到对应的分类,直接返回初始答案。接下来,将分类结果传入问题解析器进行解析,得到SQL查询语句res_sql。然后,将SQL查询语句传入答案搜索器进行搜索,得到最终的答案final_answers。最后,根据搜索结果,如果没有找到合适的最终答案,返回初始答案;否则,将最终答案连接成一个字符串,返回给用户。
在主程序文件里面的__main__中,创建了一个ChatBotGraph对象,并通过循环不断接收用户的问题,并调用chat_main方法进行回答。
当我们执行chatbot_graph.py主程序,实现知识图谱医疗问答:
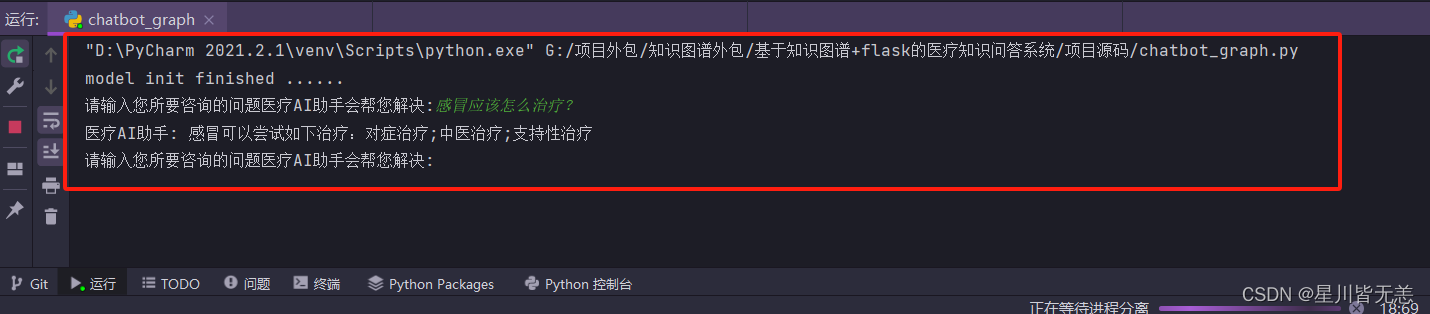
“请输入您所要咨询的问题医疗AI助手会帮您解决:”

我们输入一个简单的问题:感冒应该怎么治疗?
问答系统返回的结果如下:
再试试其它的问题:比如什么是苯中毒等等
问答系统返回结果如下:
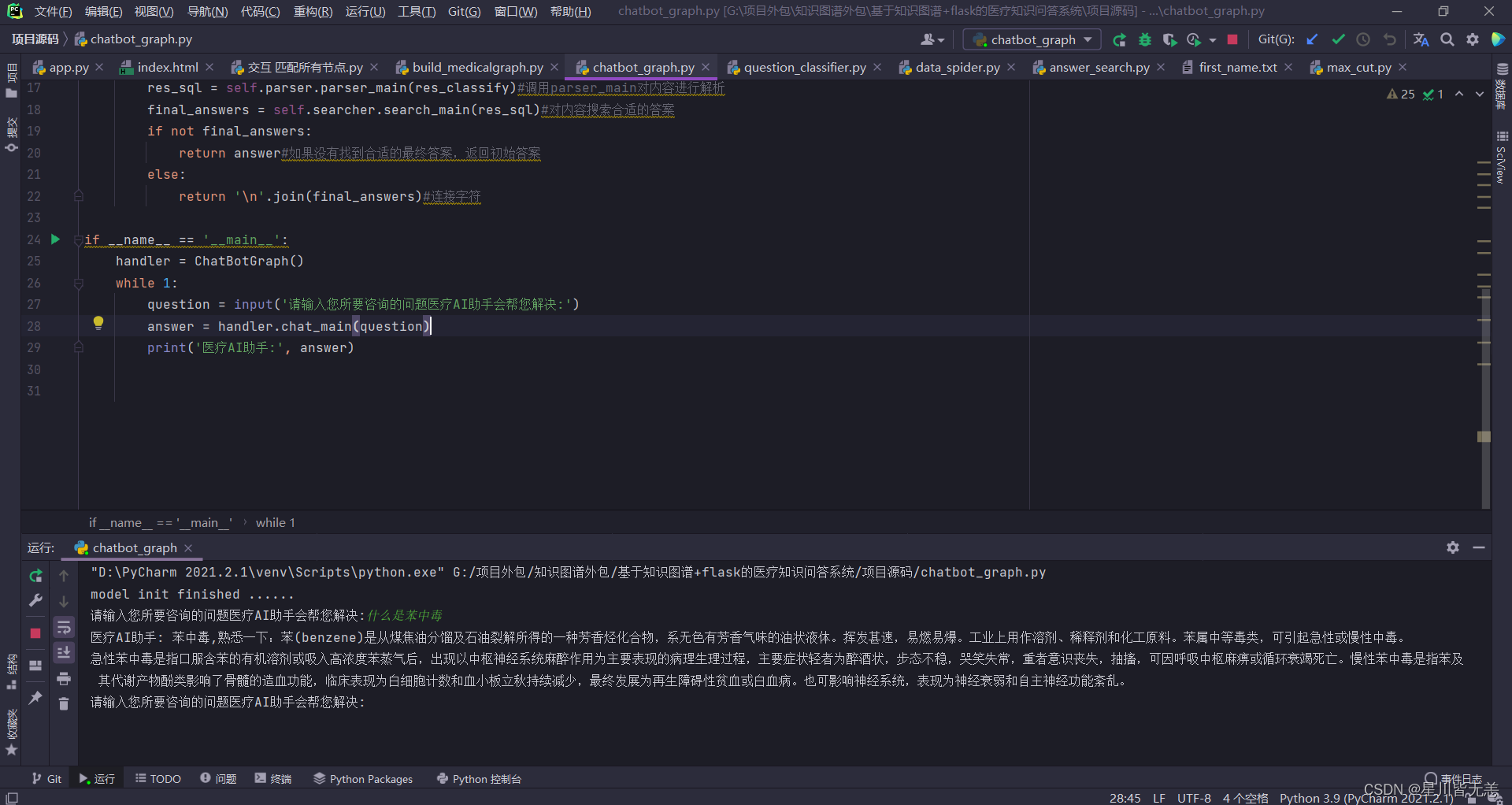
经过测试本问答系统能回答的问题有很多,基于问句中存在的关键词回答效果表现很好。做出来的基于知识图谱的医疗知识问答系统能够根据用户提出的问题很好的进行解答。
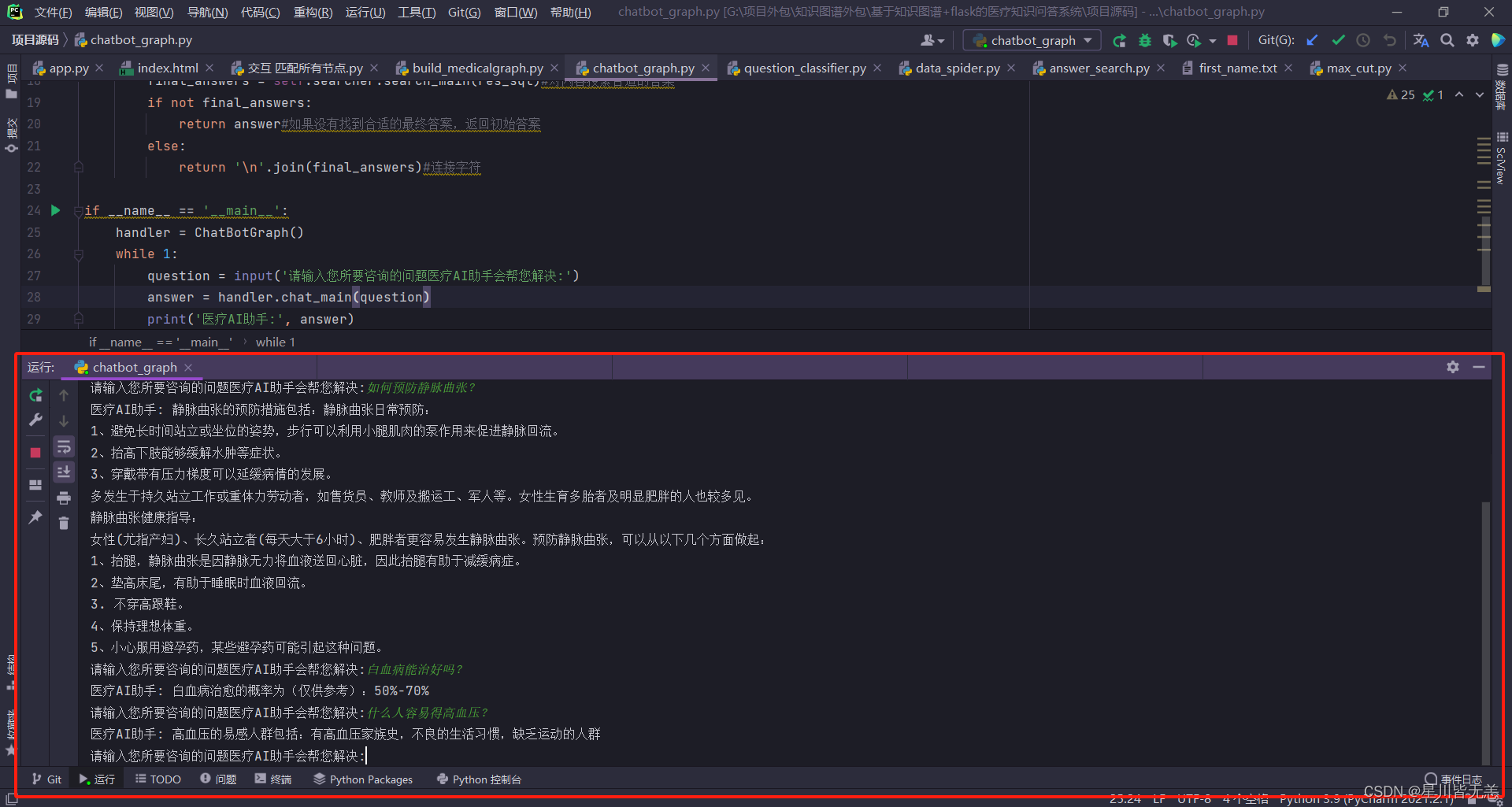
做出来的问答系统还是很Nice的。
基于很多粉丝私信我做一个简单的flask前端问答,方便一些小伙伴云平台部署。我设计了一个简单的基于 Flask 的聊天机器人应用app.py,并且用户提交的问题数据和问答系统返回的数据都会自动存储到mysql,如果想存储到其他数据库也可以自行修改。这里mysql创建表比较简单就不介绍了。
知识图谱问答系统UI设计
先导入所需模块
from flask import Flask, render_template, request import mysql.connector 创建一个Flask应用程序实例
app = Flask(__name__) 然后进行sql数据库连接,主机名、用户、mima和sql数据库名称根据实际情况自行更改。
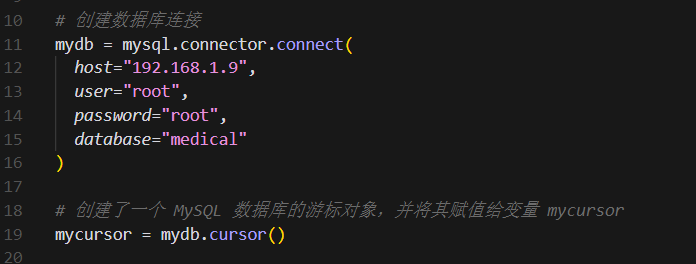
将用户输入和问答系统返回结果输出一起插入sql数据库
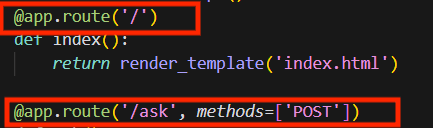
这里我设计的两个路由:
@app.route(‘/’)装饰器定义了一个路由,当用户访问根路径时,会调用index()函数并返回一个名为index.html的模板页面。
另外,@app.route(‘/ask’, methods=[‘POST’])装饰器定义了另一个路由,当用户通过POST方法提交一个表单到/ask路径时,会调用ask()函数。这个函数获取用户在表单中输入的问题,然后通过调用handler.chat_main(question)方法来获取问题的答案。接着,它将用户的问题和答案插入到一个名为chat_logs的SQL数据库表中,并将结果渲染到index.html模板页面中返回给用户
然后核心部分还是我们的ChatBotGraph 类:这是聊天机器人的核心类。init() 方法初始化了 QuestionClassifier、QuestionPaser 和 AnswerSearcher 三个类的实例。chat_main() 方法接收用户输入的问题 sent,首先使用 QuestionClassifier 对问题进行分类,然后使用 QuestionPaser 对分类结果进行解析,生成 SQL 语句,最后使用 AnswerSearcher 对 SQL 语句进行搜索,得到最终答案 final_answers。最后当我们这个脚本被直接执行时,启动 Flask 应用程序,并在调试模式下运行。如果脚本被作为模块导入,app.run(debug=True) 将不会执行,因此不会启动 Flask 服务器。
if __name__ == '__main__': app.run(debug=True) 添加了两个文件夹,里面是对flask前端页面的渲染,主要是一些静态文件的放置,如果想加入更多动态页面的设计和js逻辑可以放置到这里。

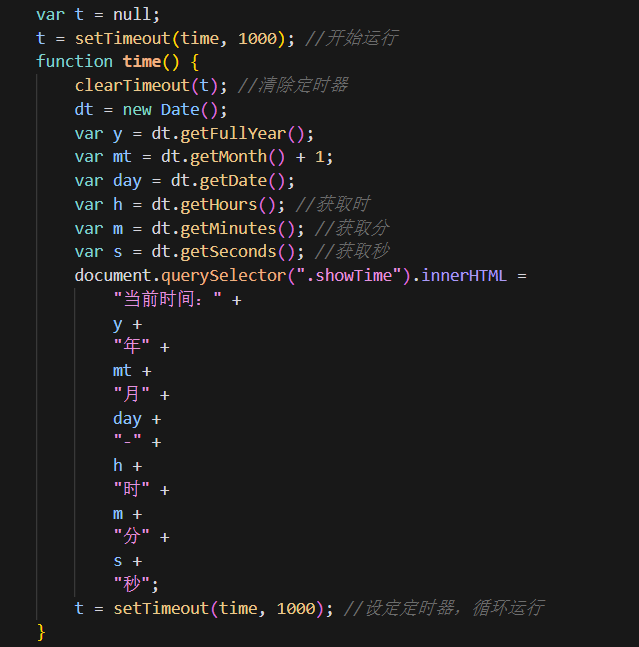
在系统页面加了一个显示时间的js逻辑代码,定义了一个名为time的函数,并使用setTimeout函数在页面加载后延迟1秒钟开始执行该函数。time函数的作用是更新页面中具有类名为showTime的元素的内容,这里我设置的是time函数每隔1秒钟执行一次,实现定时更新页面中的时间显示。
控制台启动问答系统没问题,启动我们设计的flask框架UI节目进行问答,neo4j数据库打开、mysql数据库连接没问题、问答系统所需要的包都安装,各种环境都没问题,直接启动app.py脚本

本地地址 http://127.0.0.1:5000/

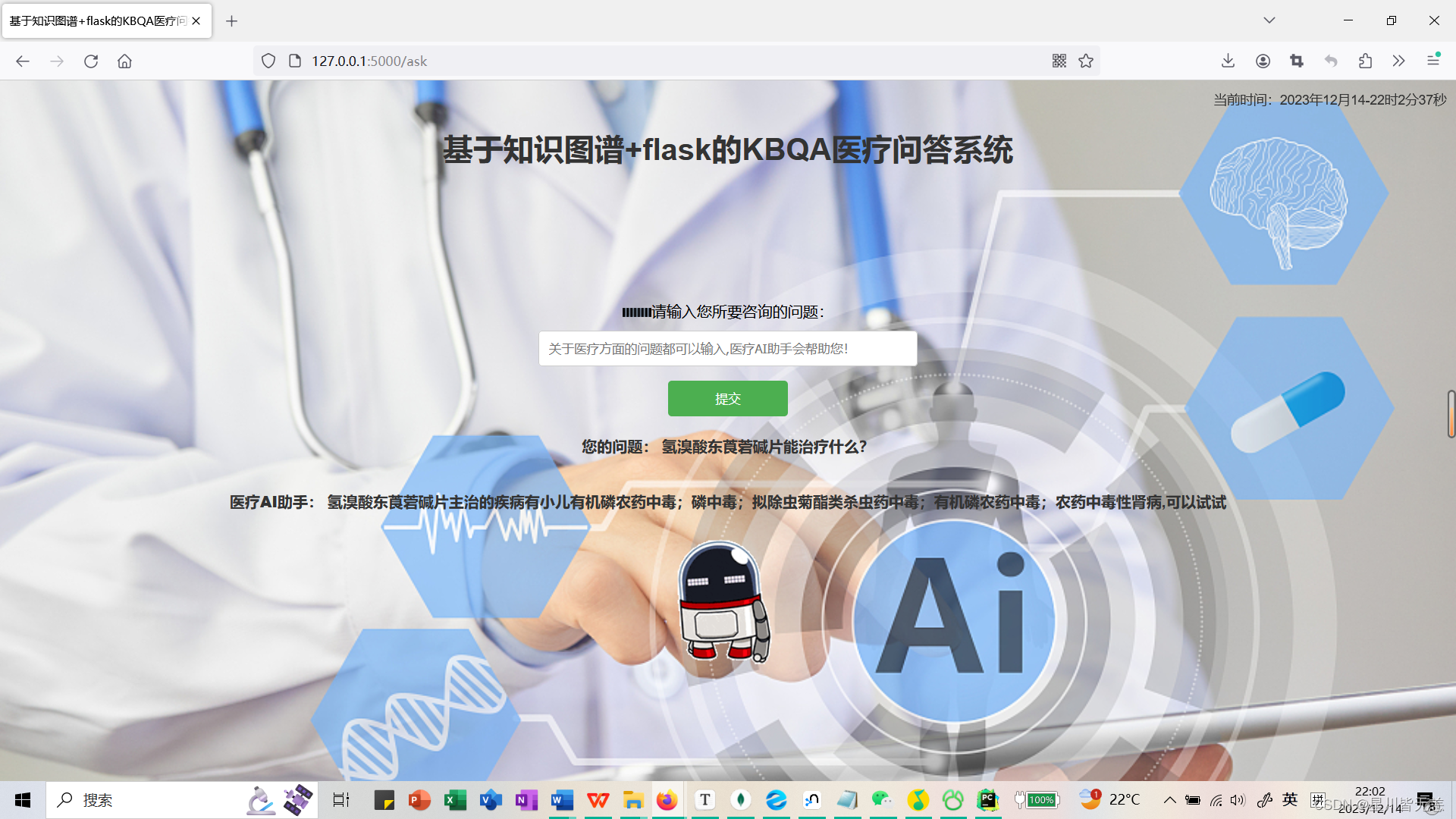
加入了动态UI设计,整体页面展示效果还是挺好的
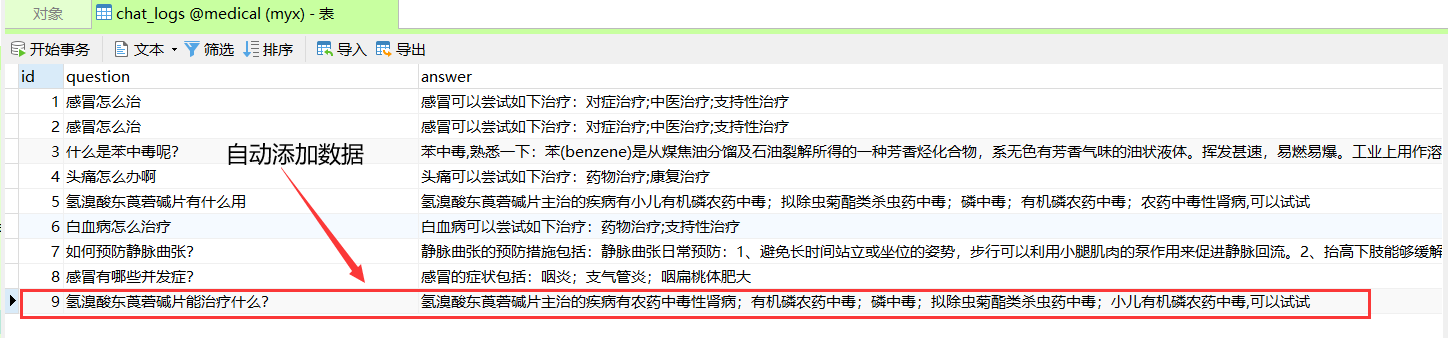
我们问一些医学上的问题,医学AI助手会进行回答,然后数据自动存储到sql数据库中的chat_logs表。
比如:“氢溴酸东莨菪碱片能治疗什么?”

医疗AI助手返回回答结果
数据库中的表数据也是自动添加。
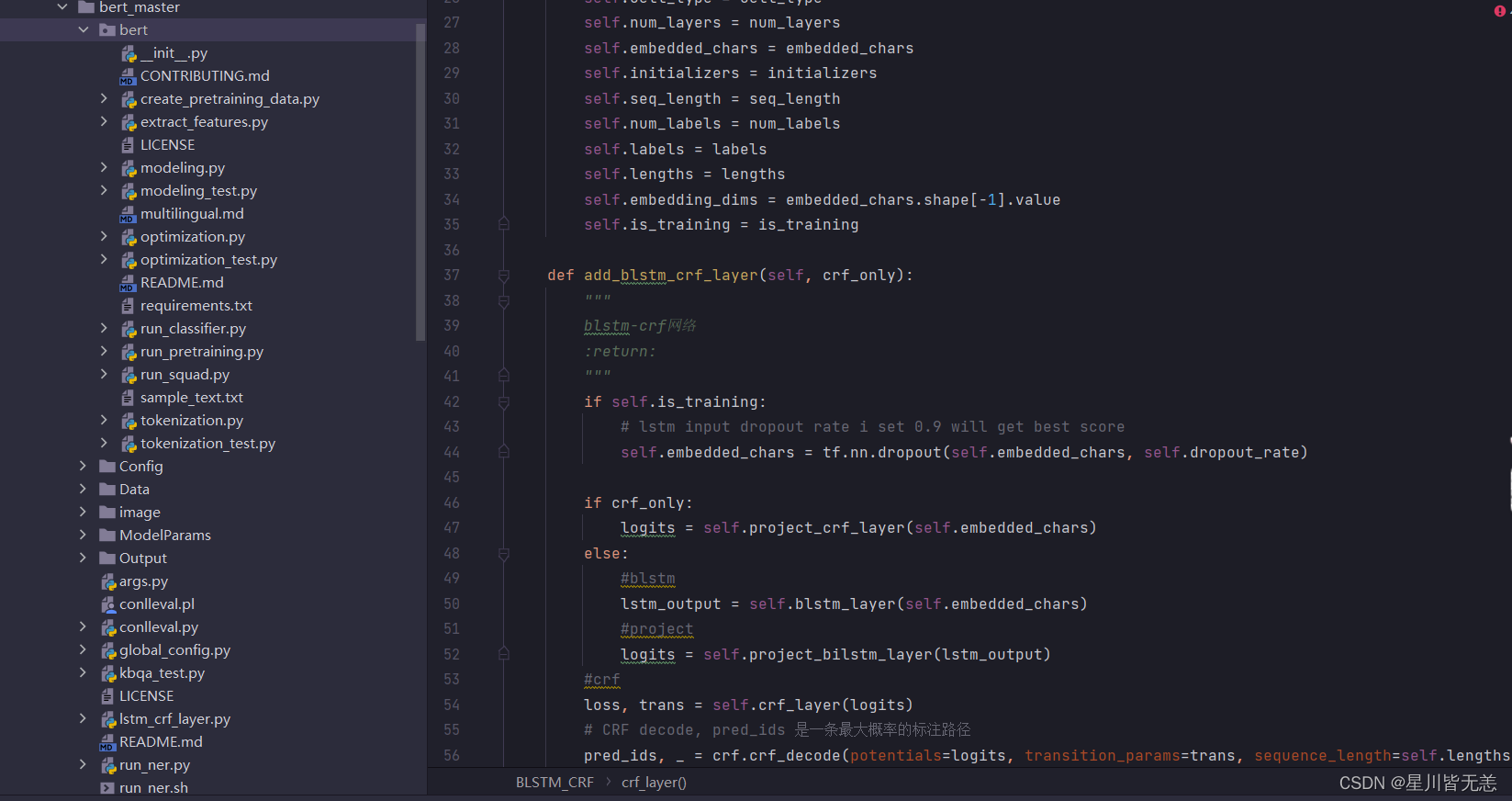
十、封装深度学习完整系统(非开源)——基于BERT+LSTM+CRF深度学习识别模型医疗知识图谱问答可视化系统
BERT是一种基于Transformer 架构的预训练语言模型,能够捕捉双向上下文信息。BERT 模型在大规模语料上进行预训练,然后可以通过微调来适应特定任务,BERT 可用于处理输入文本,提取丰富的语义信息。它可以用于文本的编码和表征学习,以便更好地理解医学问答中的问题和回答。LSTM 是一种递归神经网络(RNN)的变体,专门设计用于处理序列数据。它通过使用门控机制来捕捉长期依赖关系,适用于处理时间序列和自然语言等序列数据。 LSTM 可以用于处理医学文本中的序列信息,例如病历、症状描述等。它有助于保留文本中的上下文信息,提高模型对长文本的理解能力。CRF 是一种用于标注序列数据的统计建模方法。在序列标注任务中,CRF 能够考虑标签之间的依赖关系,从而更好地捕捉序列结构。 在医学文本中,CRF 可以用于命名实体识别(NER)任务,例如识别疾病、药物、实验室结果等实体。通过引入CRF层,可以提高标签之间的一致性和整体序列标注的准确性。
部分代码,其它内容就不再过多描述。
具体详情见另一篇文章:大数据知识图谱之深度学习——基于BERT+LSTM+CRF深度学习识别模型医疗知识图谱问答可视化系统
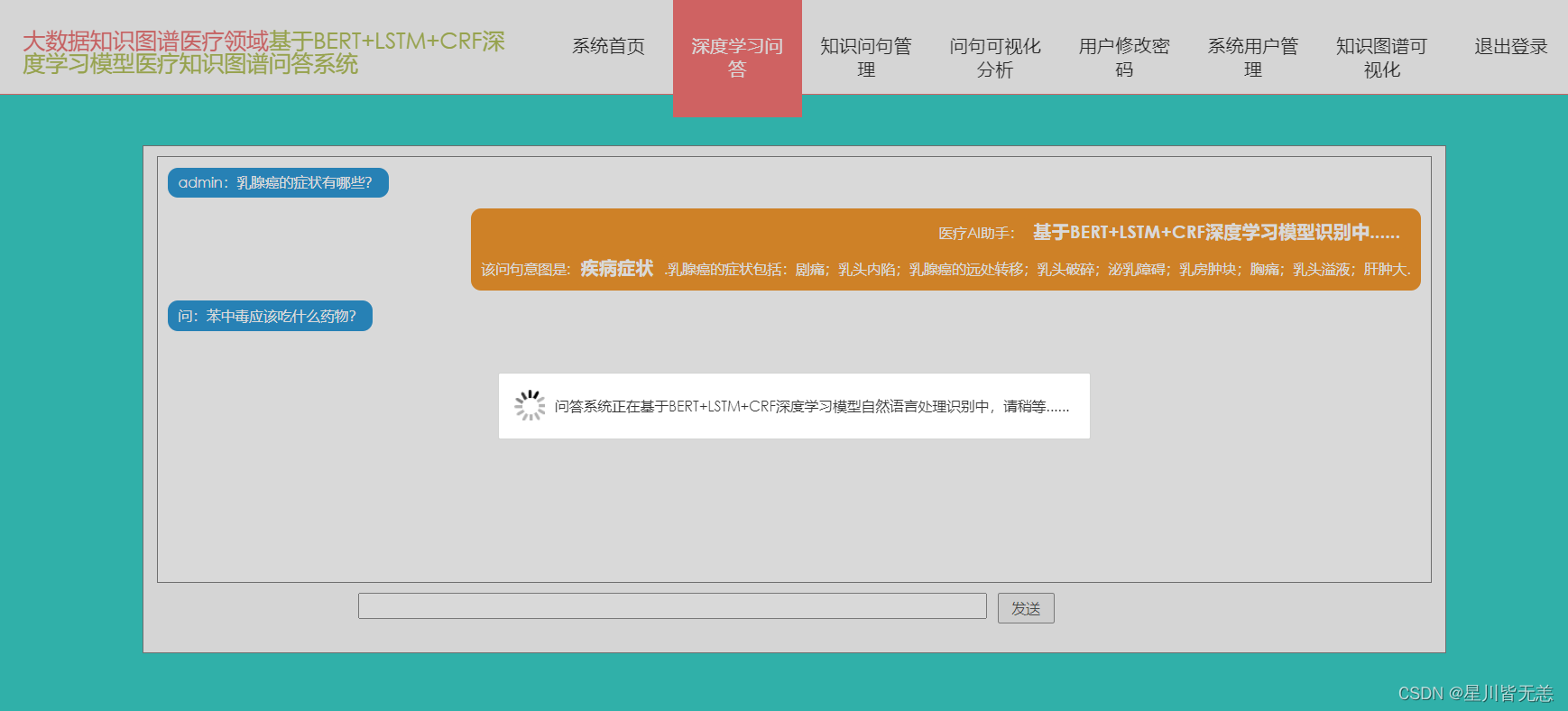
核心深度学习问答界面:
此次基于BERT+LSTM+CRF深度学习识别模型医疗知识图谱问答可视化系统的主要功能模块为深度学习问答模块,用户可以通过该功能模块实现深度学习问答,通过界面下方的输入栏实现医疗领域相关问题的录入,通过点击发送实现提问,系统会结合用户端的问题进行意图分析并反馈问题答复内容,具体深度学习问答界面的主要功能栏内容呈现如下。
部分系统截图展示:
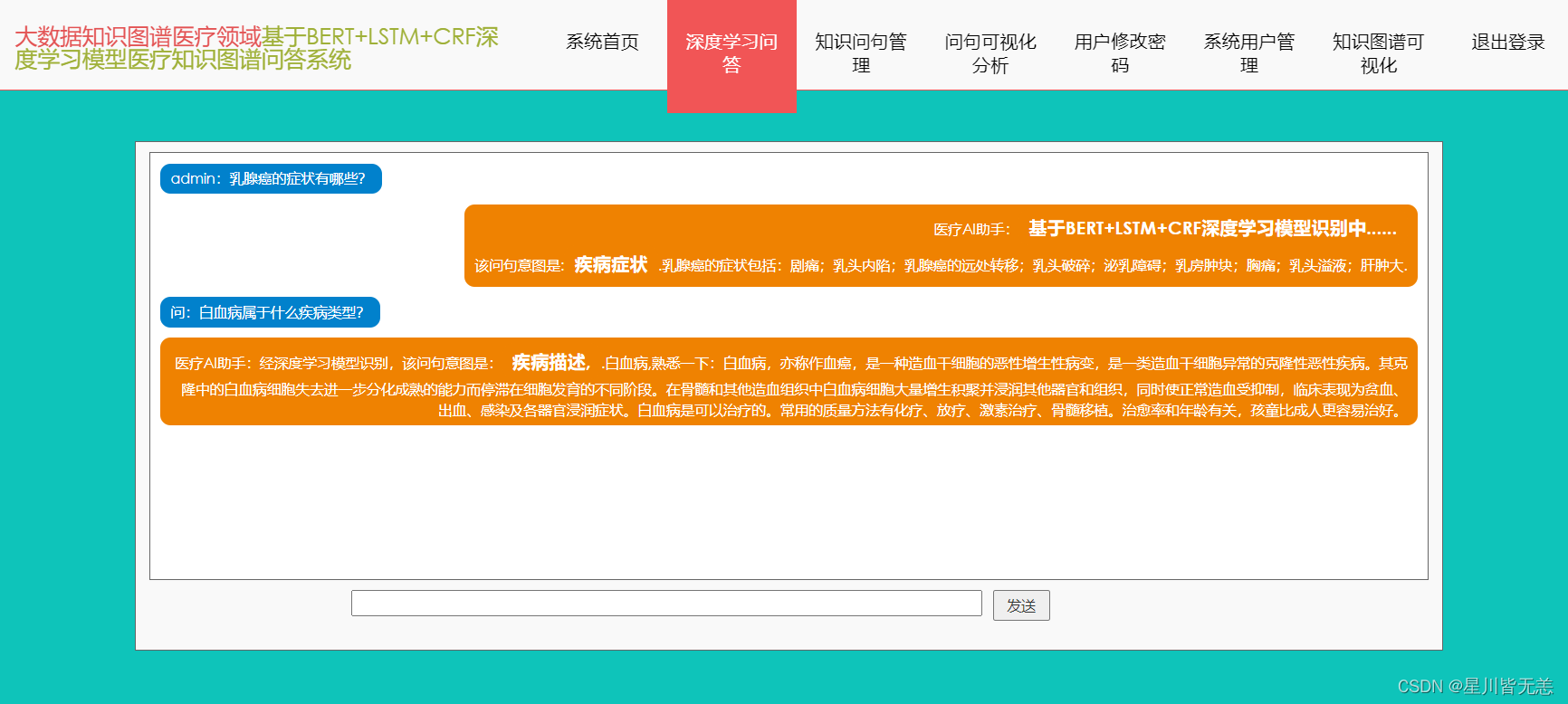
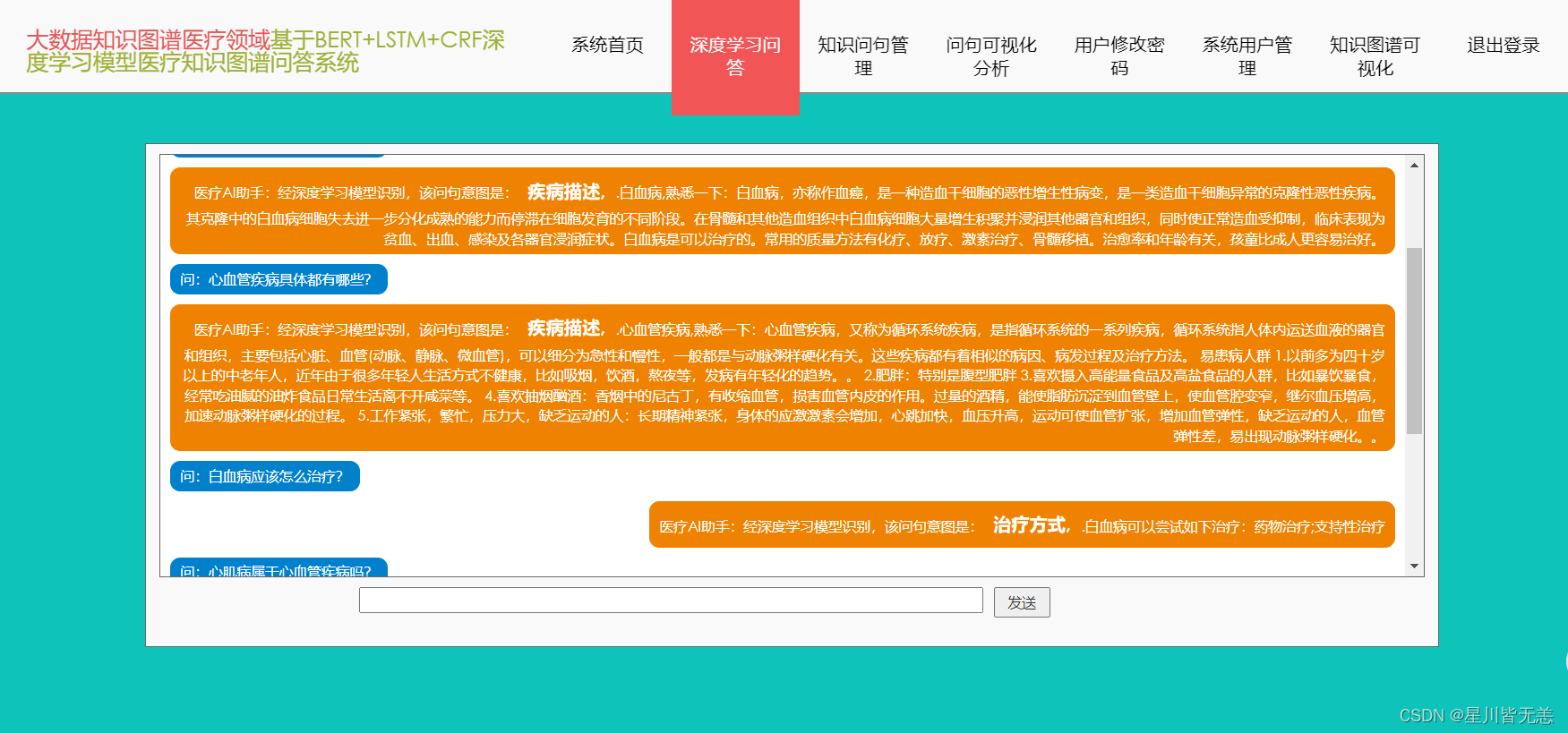
其它详情不再过多描述,感兴趣的可以扫码/私信探讨学习交流。
十一、用Python实现增删改查 数据查询及维护
用Python实现增加节点
过于简单不再多说。
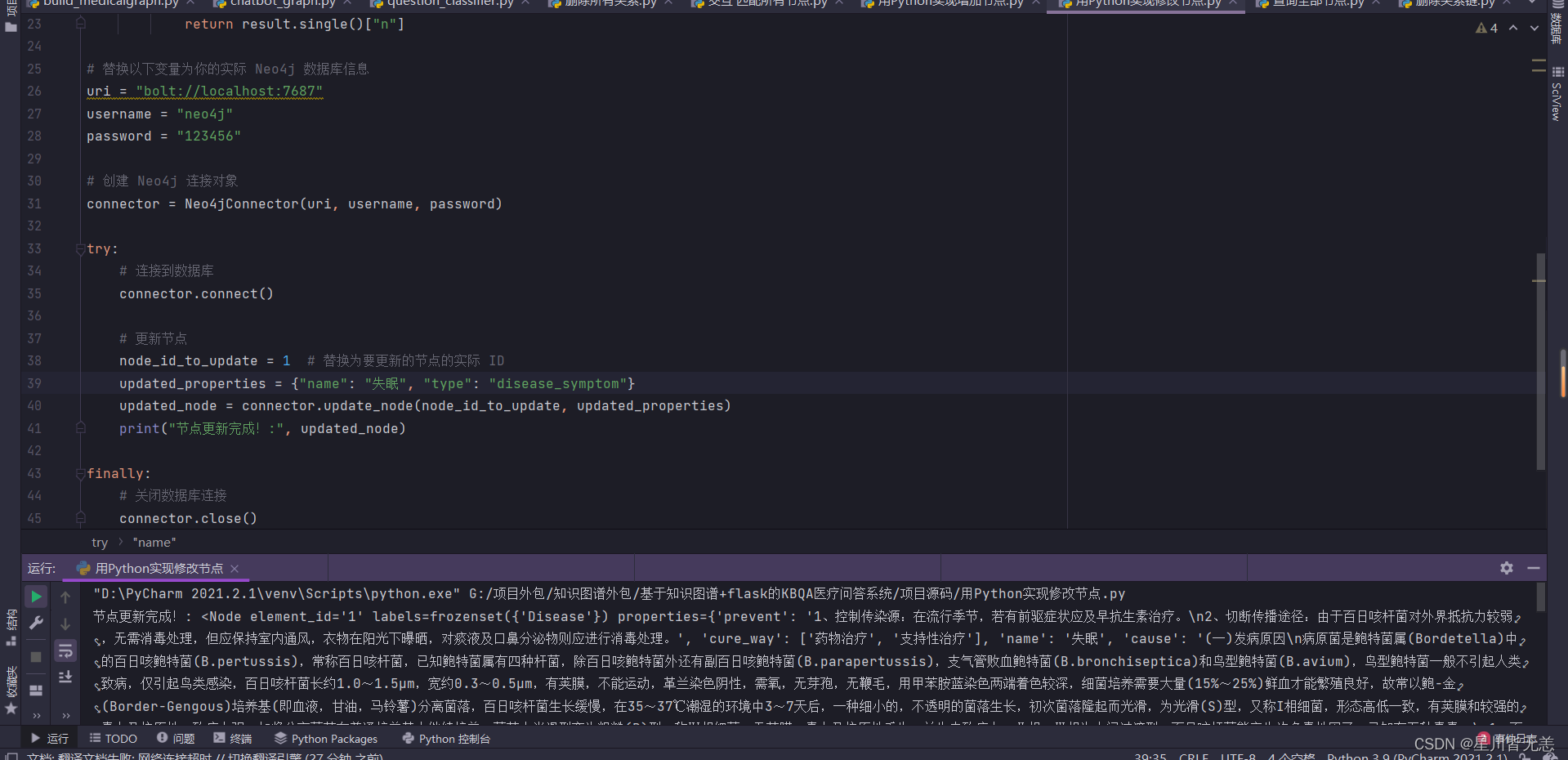
用Python实现修改节点
update_node 方法用于更新节点。它接收要更新的节点的 ID 和新的属性作为参数,并返回更新后的节点。你可以将 node_id_to_update 更改为你想要更新的节点的实际 ID,将 updated_properties 更改为你想要设置的新属性,然后调用 update_node 方法即可修改节点。
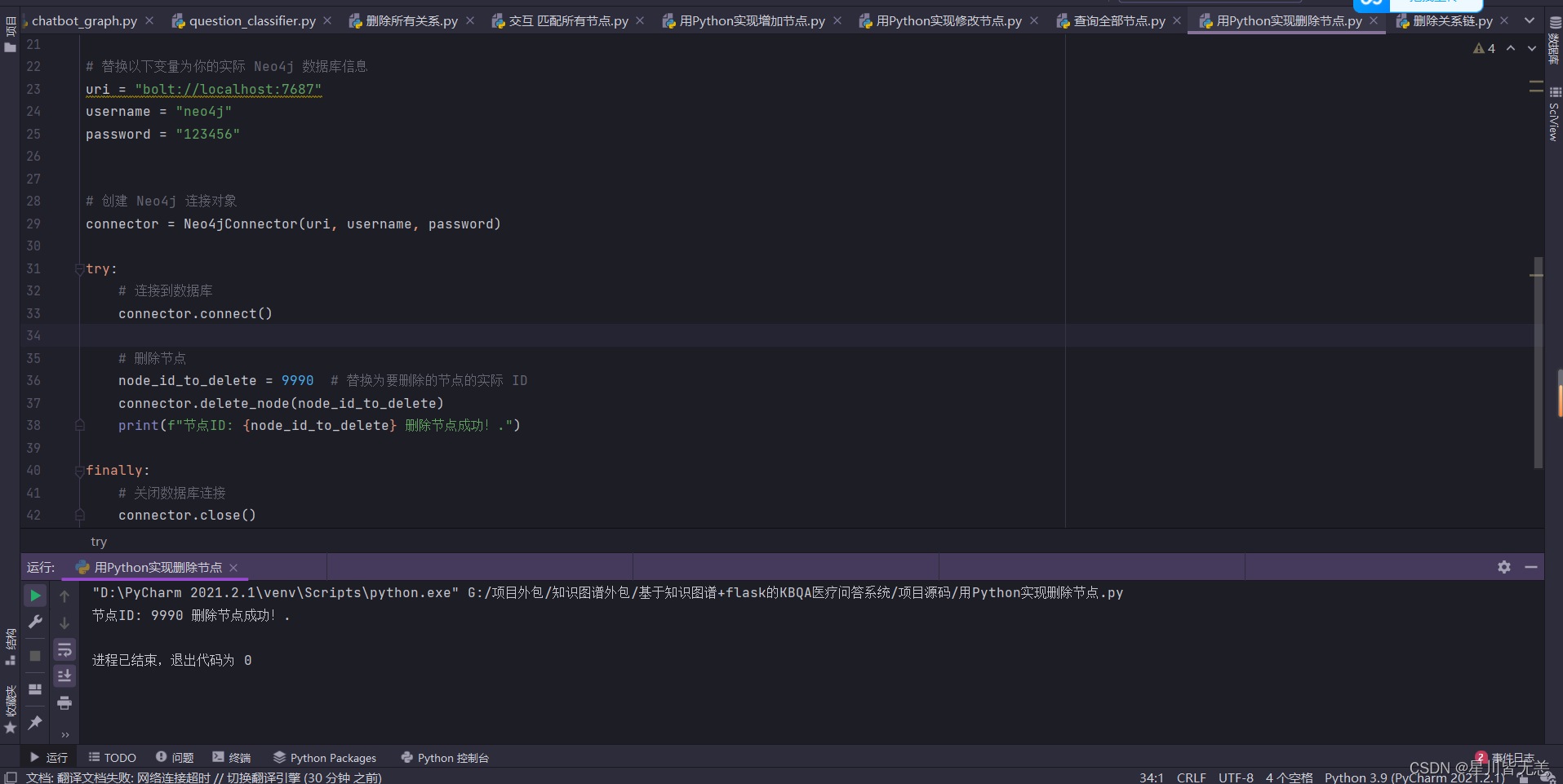
用Python实现删除节点
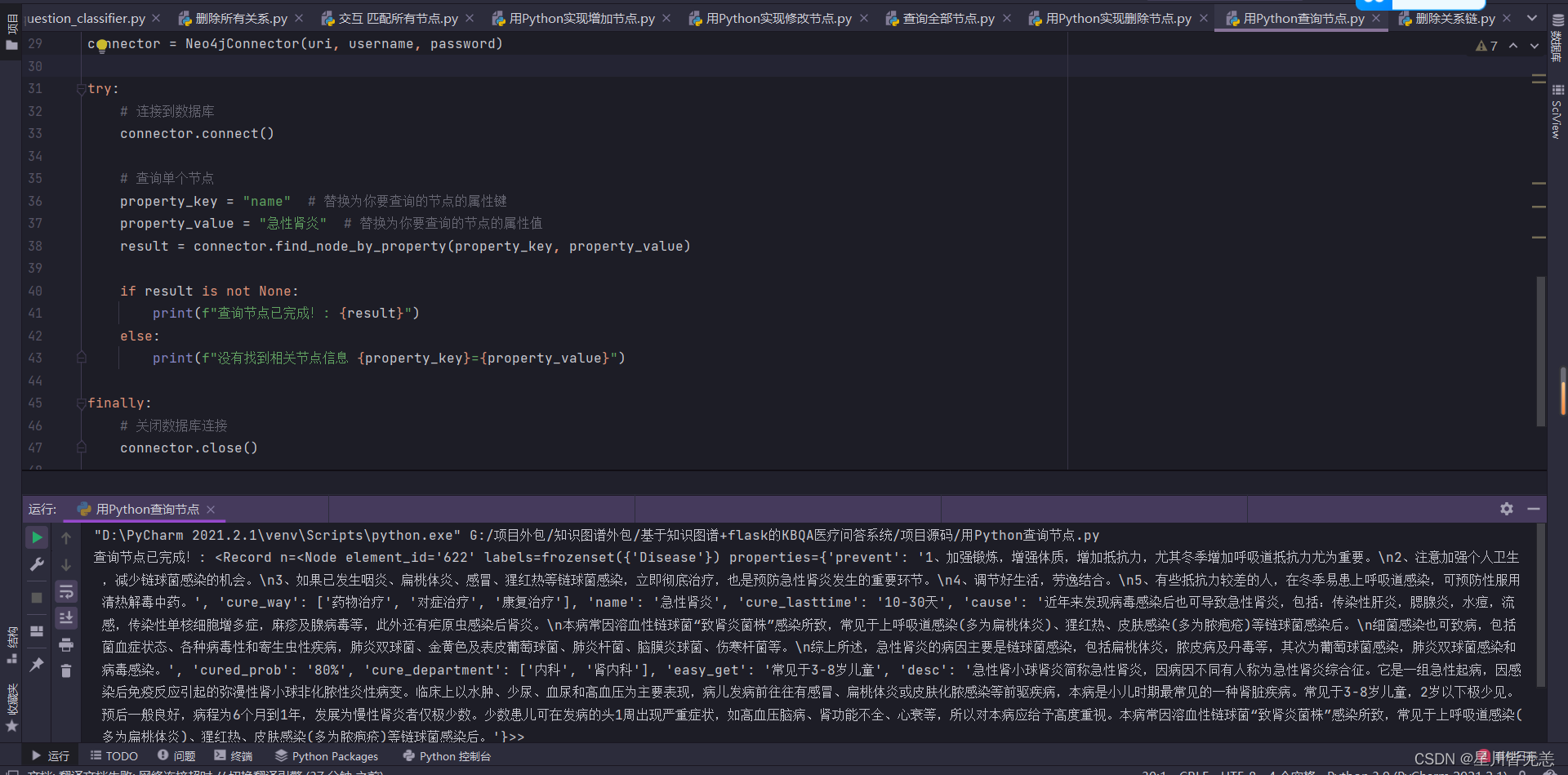
用Python实现查询节点
find_node_by_property 方法接收节点属性的键和值作为参数,然后执行一个 Cypher 查询,查找具有指定属性的节点。查询使用 MATCH 子句和 WHERE 子句来过滤节点,然后返回符合条件的节点。
参考资料:
https://zhuanlan.zhihu.com/p/683241607
https://news.sohu.com/a/720270336_121124371
参考文献:
陈明,刘蓉,熊回香.基于医疗知识图谱的智能问答系统研究[J/OL].情报科学,1-9[2024-04-13].
黄涌,葸娟霞,关成斌.基于BERT-BiGRU模型的智慧医疗问答系统[J].软件工程,2024,27(03):11-14+25.
十一、结语
需项目源码文档解析等资料/解析/商业合作/交流探讨~等可以评论留言/添加下面个人名片,感谢各位的喜欢与支持!
后面有时间和精力也会分享更多关于大数据领域方面的优质内容,喜欢的小伙伴可以点赞收藏加关注,感谢各位的喜欢与支持
