2024甲辰龙年2月22日始
碎碎念:生信小白一枚,去年暑假就打算开始坚持写博文的,一直到现在才开始,拖延症属实晚期了!不过只要开始就还不算晚。
记录自己每天做的学的东西,目的是督促自己(其实是记性差)希望自己能坚持下去!
最近在研究Alphafold/Alphafold-Multimer的使用,第一步当然是找教程学习在服务器上安装(没有可用服务器的小伙伴可以使用谷歌的Colab试试)。废话不多说,下面是我根据教程一步一步的安装过程:
1、conda创建alphafold2的虚拟环境并激活
conda create --name alphafold python==3.8 conda activate alphafold mkdir Alphafold #建个文件夹,后面存放alphafold的各种文件2、安装所需依赖
conda install -y -c conda-forge openmm==7.5.1 cudnn==8.2.1.32 cudatoolkit==11.0.3 pdbfixer==1.7 conda install -y -c bioconda hmmer==3.3.2 hhsuite==3.3.0 kalign2==2.04 pip install absl-py==1.0.0 biopython==1.79 chex==0.0.7 dm-haiku==0.0.9 dm-tree==0.1.6 immutabledict==2.0.0 jax==0.3.25 ml-collections==0.1.0 numpy==1.21.6 pandas==1.3.4 protobuf==3.20.1 scipy==1.7.0 tensorflow-cpu==2.9.0 pip install --upgrade jax==0.3.25 jaxlib==0.3.25+cuda11.cudnn82 -f https://storage.googleapis.com/jax-releases/jax_cuda_releases.html 3、下载Alphafold V2.2.4
最开始下载的是V2.3.0,但是后面安装依赖openmm patch出错,查了一下说是2.3.0版本之后docker文件里就没有openmm.patch这个文件了。
后续补充:2.3版本及以上没有补丁解决方法:手动打补丁!参照这篇PSP - 关于 AlphaFold2 的 openmm.patch 补丁_patch -p0 < $alphafold_path/docker/openmm.patch-CSDN博客
找到site-packages里的simtk/openmm/app/topology.py文件,按照已经打过补丁的文件,自己对照着修改就行,亲测可用!
下载方法一:直接下载或是git克隆方法我没用过,不知道咋样。不过这样下载,后面想更新代码的话就可以直接使用官方命令git fetch origin main。
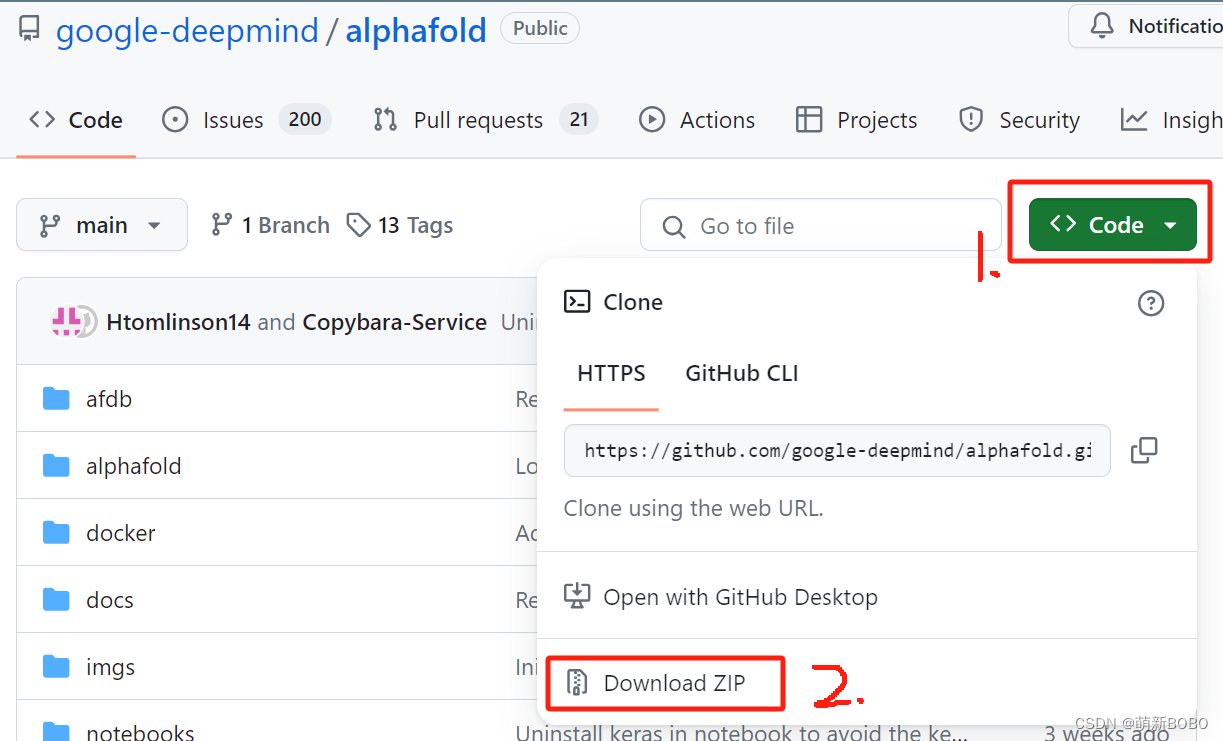
wget https://github.com/deepmind/alphafold/archive/refs/tags/v2.3.1.tar.gz && tar -xzf v2.3.1.tar.gz && export alphafold_path="$(pwd)/alphafold-2.3.1" git clone https://github.com/deepmind/alphafold.git下载方法二:我用的。在github找到要下载的版本,按下图这样把文件下载好,我是用FileZilla传到服务器里的前面建的Alphafold文件夹里,就算下载好了。该方法后续更新代码使用git fetch origin main会报错,我自己又找方法设置了一下,还是没成功。
设置一下alphafold_path路径 alphafold_path="/home/用户名/Alphafold"4、下载化学性质(chemical properties)到alphafold里的common文件夹内
cd common/ wget -q -P $alphafold_path/alphafold/common/ https://git.scicore.unibas.ch/schwede/ope/raw/7102c63615b64735c4941278d92b554ec94415f8/modules/mol/alg/src/stereo_chemical_props.txt后续补充:最开始都没发现根本就没下载到stereo_chemical_props.txt这个文件,直到运行报错才发现。不过在github上找到了别人的解决办法:将-q 换成 --no-check-certificate 就可以了。
wget --no-check-certificate -P alphafold/alphafold/common/ https://git.scicore.unibas.ch/schwede/openstructure/-/raw/7102c63615b64735c4941278d92b554ec94415f8/modules/mol/alg/src/stereo_chemical_props.txt5、添加openmm.patch补丁
cd /home/用户名/anaconda3/envs/alphafold/lib/python3.8/site-packages/ && patch -p0 < $alphafold_patnmm.patch6、修改run_alphafold.sh的权限并运行,不报错就是安装成功了。
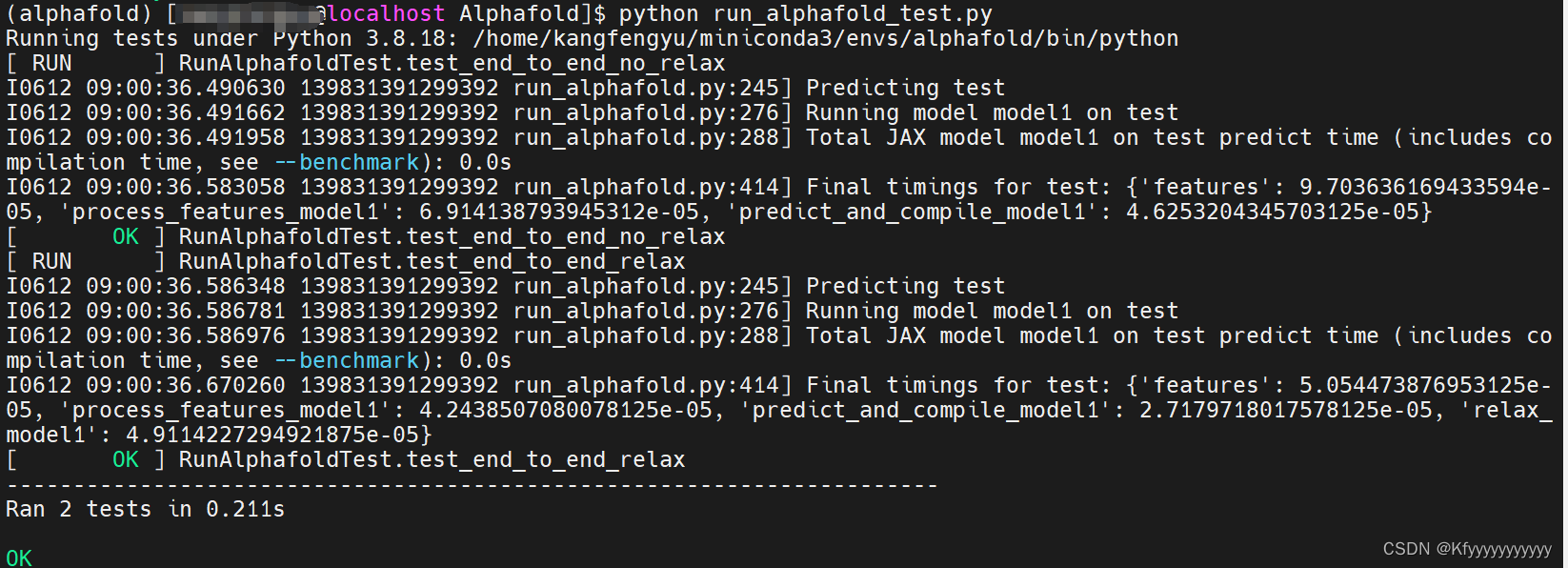
cd Alphafold/ chmod 777 run_alphafold.sh bash run_alphafold.sh python run_alphafold_test.py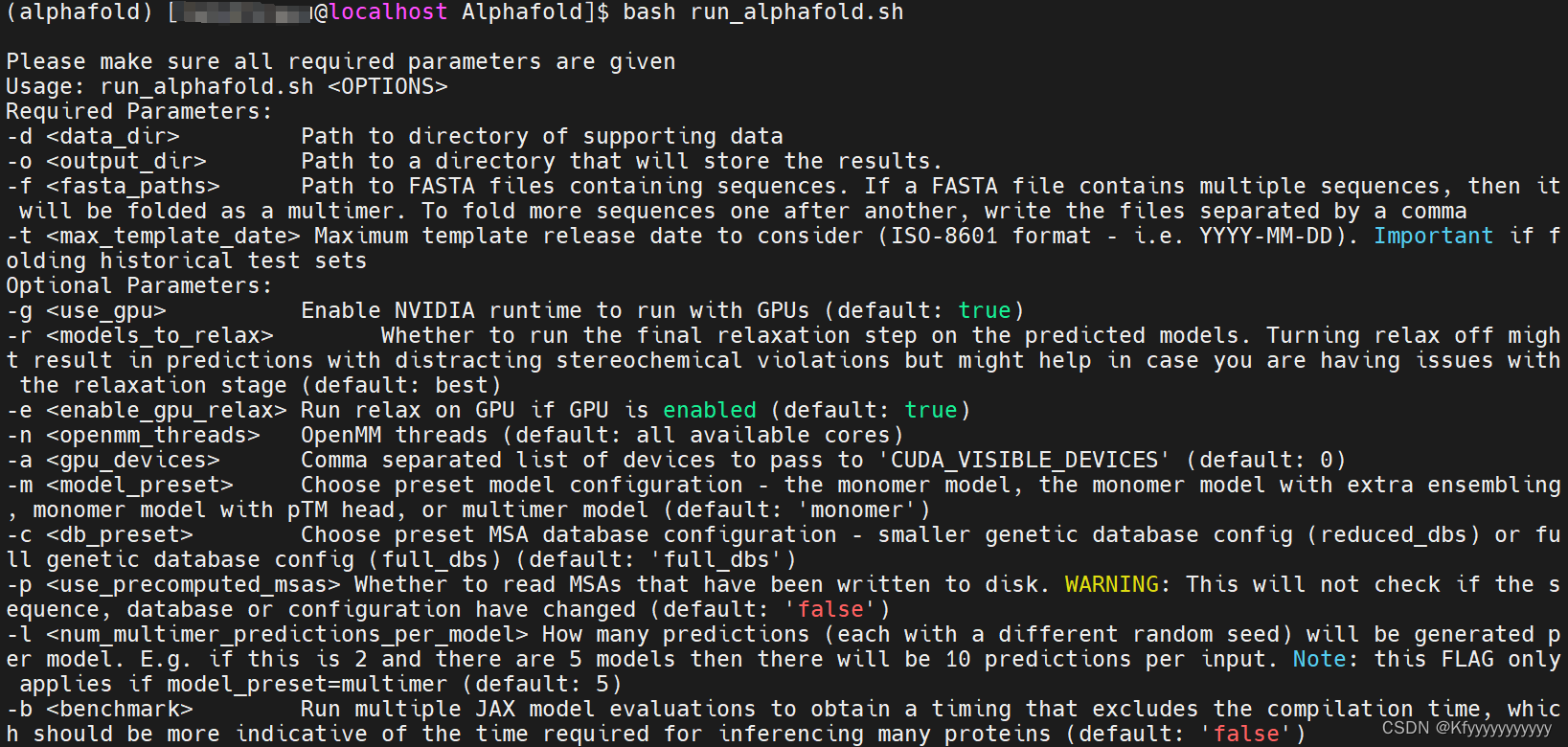
 7、下载数据集(pdb_mmcif需要用另外的方法!)
7、下载数据集(pdb_mmcif需要用另外的方法!)
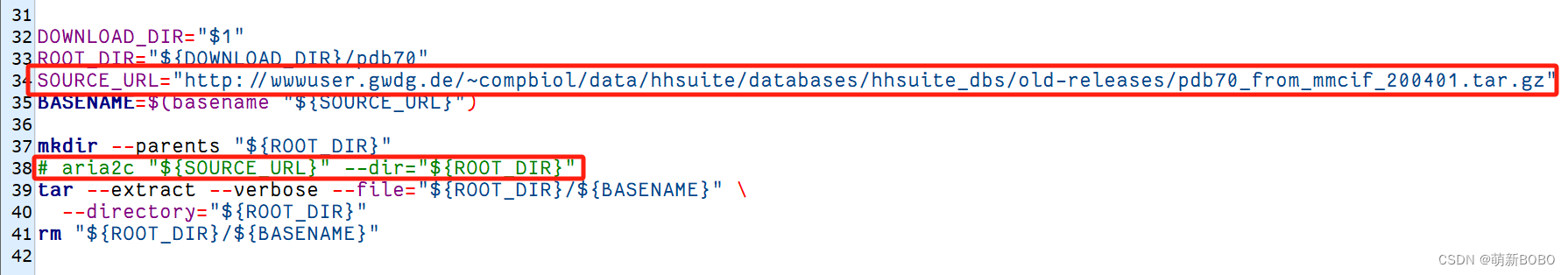
这是最麻烦的过程。不要直接运行download_all_data.sh,下载所有数据,大概率是不成功的,虽然我没试。我是使用IDM下载器手动一个一个下载的。打开scripts文件夹内的随便一个脚本,找到下载地址,复制到IDM上下载就可以了。然后传到服务器上的dataset文件夹里的相应数据库文件夹(给每个数据库都建个文件夹)。SOURCE_URL后面就是下载链接。脚本里是用aria2c下载的,没有的需要先安装,使用conda就可以。因为我们已经下载好了,所以把这行注释掉就可以了(下图,加个#就注释掉了),只解压就可以了。
bash scripts/download_alphafold_params.sh /home/用户名/Alphafold/alphafold/dataset 8、pdb_mmcif数据集的下载
这个比较麻烦,不能用7中的方法。这个数据集使用了同步(rsync)的方式进行下载。好多人推荐根据rsync 多进程并发执行同步数据 - 技术写真 - 若海的方法进行多线程同步,代码如下:
#!/bin/sh src='rsync.rcsb.org::ftp_data/structures/divided/mmCIF' #源路径,结尾不带斜线 dst='./pdb_mmcif/raw' #目标路径,结尾不带斜线 opt="--recursive --links --perms --times --compress --info=progress2 --delete --port=33444" #同步选项 num=10 #并发进程数 depth='5 4 3 2 1' #归递目录深度 task=/tmp/`echo $src$ | md5sum | head -c 16` [ -f $task-next ] && cp $task-next $task-skip [ -f $task-skip ] || touch $task-skip # 创建目标目录结构 rsync $opt --include "*/" --exclude "*" $src/ $dst # 从深到浅同步目录 for l in $depth ;do # 启动rsync进程 for i in `find $dst -maxdepth $l -mindepth $l -type d`; do i=`echo $i | sed "s#$dst/##"` if `grep -q "$i$" $task-skip`; then echo "skip $i" continue fi while true; do now_num=`ps axw | grep rsync | grep $dst | grep -v '\-\-daemon' | wc -l` if [ $now_num -lt $num ]; then echo "rsync $opt $src/$i/ $dst/$i" >>$task-log rsync $opt $src/$i/ $dst/$i & echo $i >>$task-next sleep 1 break else sleep 5 fi done done done 但是我执行该脚本时,可能是因为我们学校网络的原因,同步下载的每一项都无法达到100%,担心后面运行出错,我就放弃了这种多线程的方法。就直接使用了官方自带的脚本,也是同步(rsync)的方式进行下载。执行下面的命令:断断续续下了有1天吧,我们学校的网最近限速,最高才3M/s,服务器也总是会断。查了一下rsync是默认会中断续传的,下载自动解压后数据库有275G,也不知道是不是完整的,到时候运行看看会不会报错。obsolete.dat这个文件如果无法下载,就复制他的下载地址下载,然后再传到服务器pdb_mmcif文件夹里就行了。
bash ./scripts/download_pdb_mmcif.sh /home/用户名/Alphafold/dataset官方贴出的所需数据库的信息:数据库大小会有更新,应该只会比这个大,不会小。
$DOWNLOAD_DIR/ # Total: ~ 2.62 TB (download: 556 GB) bfd/ # ~ 1.8 TB (download: 271.6 GB) # 6 files. mgnify/ # ~ 120 GB (download: 67 GB) mgy_clusters_2022_05.fa params/ # ~ 5.3 GB (download: 5.3 GB) # 5 CASP14 models, # 5 pTM models, # 5 AlphaFold-Multimer models, # LICENSE, # = 16 files. pdb70/ # ~ 56 GB (download: 19.5 GB) # 9 files. pdb_mmcif/ # ~ 238 GB (download: 43 GB) mmcif_files/ # About 199,000 .cif files. obsolete.dat pdb_seqres/ # ~ 0.2 GB (download: 0.2 GB) pdb_seqres.txt small_bfd/ # ~ 17 GB (download: 9.6 GB) bfd-first_non_consensus_sequences.fasta uniref30/ # ~ 206 GB (download: 52.5 GB) # 7 files. uniprot/ # ~ 105 GB (download: 53 GB) uniprot.fasta uniref90/ # ~ 67 GB (download: 34 GB) uniref90.fasta我是根据很多教程一点一点安装下载的,完了之后才写的这篇,导致中间一些过程没有截图,写的也不够完全。加上的后续补充是我运行时出现的问题,找到了解决办法觉得还是放在这里比较好!
这类的教程非常多,感谢各位大佬的分享,让我这种小白也能一点一点完成。写这些主要是记录一下我的学习过程,防止遗忘,能时常翻来看看,如果恰巧也能帮到你,那真是太好了!如果哪里有错误的地方,请各位大佬多多指出来!后续的运行过程,我也会记录下来,希望自己能坚持下去!2024年2月29日最后贴上一张很有意思的图: